# Instalación desde R.
install.packages("dlookr", dependencies = T)
# Versión de desarollo
# devtools::install_github("choonghyunryu/dlookr")
# Si no puede usar R o Rstudio, puede recurrirse al servicio de Posit en la nube:
# https://posit.cloud/Radiografía de datos con dlookr: más allá del summary().
Limpia, fija y da esplendor
1 Programador y mantenimiento
![]()
- Nombre: Choonghyun Ryu
- Contacto: choonghyun.ryu@gmail.com
- GitHub: choonghyunryu/dlookr
- Origen: El término dlookr sintetiza “looking at the data”.
- Versión 0.6.3 publicada en CRAN
- Fecha: 7 de febrero de 2024
2 Introducción a la librería dlookr de R
- dlookr es una colección de herramientas que trata de facilitar el diagnóstico, exploración y transformación de datos.
- Ofrece información y visualizaciones de valores faltantes, valores atípicos, valores únicos y negativos, etc., ayudando a entender la distribución y calidad de los datos.
- Más información sobre la librería en: https://cran.r-project.org/web/packages/dlookr/index.html
2.1 Diagnostica, explora…
- Diagnóstico:
- Datos categóricos.
- Numéricos.
- Outliers.
- Valores perdidos.
- Explora:
- Estadísticos descriptivos.
- Comprobación de la normalidad.
- Comprobación de la asimetría (skewness).
- Correlación.
2.2 Diagnostica, explora y repara
- Imputación en datos perdidos numéricos:
- Media.
- Mediana.
- Moda.
- knn.
- Imputación en datos perdidos categóricos:
- Moda.
- rpart (recursive partitioning and regression trees).
- mice (multivariate imputation by chained equations).
2.3 Diagnostica, explora y repara
- Imputación en outliers:
- Media.
- Mediana.
- Moda.
- Capado.
- Agrupamiento (binning).
2.4 Instalación
2.5 Cargando paquetes
library(tidyverse)
library(dlookr)
library(readr) # Para leer los datos
library(flextable) # No necesario
library(gridExtra) # No necesario2.6 Dataset de ejemplo
Una historia de nuestros datos:
El cuestionario CUDAD trata de recoger datos sobre la actitud del alumnado de Educación a la formación de datos y a las asignaturas de metodología de investigación en Educación.
Como es típico (en CCSS) está lleno de variables ordinales y casi ninguna variable escalar.
El dataset está disponible en: Repositorio Gitlab
dfm <- read_delim("https://gitlab.com/amataste/dlookr_sesion_grupormalaga/-/raw/main/dataset_ejercicios.csv",
delim = "\t"
)
# View(dfm)3 Diagnóstico
3.1 Sintaxis para el diagnóstico
Diagnóstico de los datos:
- diagnose(dataset)
- diagnose(dataset, variable1, variable2,…) o alternativamente -variable
- Las variables se pueden indicar por el nombre o por la posición en el dataset
3.2 Sintaxis para el diagnóstico: general
diagnose(dfm)# A tibble: 39 × 6
variables types missing_count missing_percent unique_count unique_rate
<chr> <chr> <int> <dbl> <int> <dbl>
1 N numeric 0 0 243 1
2 Universidad character 0 0 3 0.0123
3 Sexo character 2 0.823 3 0.0123
4 Edad numeric 5 2.06 14 0.0576
5 Estudios character 2 0.823 6 0.0247
6 Curso numeric 0 0 5 0.0206
7 V1 numeric 6 2.47 7 0.0288
8 V2 numeric 57 23.5 7 0.0288
9 V3 numeric 34 14.0 7 0.0288
10 V4 numeric 0 0 6 0.0247
# ℹ 29 more rows3.3 Sintaxis para el diagnóstico: algunas variables
diagnose(dfm, 2, 3, 7:10)# A tibble: 6 × 6
variables types missing_count missing_percent unique_count unique_rate
<chr> <chr> <int> <dbl> <int> <dbl>
1 Universidad character 0 0 3 0.0123
2 Sexo character 2 0.823 3 0.0123
3 V1 numeric 6 2.47 7 0.0288
4 V2 numeric 57 23.5 7 0.0288
5 V3 numeric 34 14.0 7 0.0288
6 V4 numeric 0 0 6 0.02473.4 Sintaxis para el diagnóstico: por grupos
Por grupos, complementándose con dplyr
dfm %>%
group_by(Sexo) %>%
diagnose(2, 7:10)# A tibble: 15 × 8
variables types Sexo data_count missing_count missing_percent unique_count
<chr> <chr> <chr> <int> <dbl> <dbl> <int>
1 Universidad char… F 213 0 0 3
2 Universidad char… M 28 0 0 3
3 Universidad char… <NA> 2 0 0 1
4 V1 nume… F 213 5 2.35 7
5 V1 nume… M 28 1 3.57 6
6 V1 nume… <NA> 2 0 0 1
7 V2 nume… F 213 49 23.0 7
8 V2 nume… M 28 8 28.6 6
9 V2 nume… <NA> 2 0 0 1
10 V3 nume… F 213 31 14.6 7
11 V3 nume… M 28 3 10.7 7
12 V3 nume… <NA> 2 0 0 2
13 V4 nume… F 213 0 0 6
14 V4 nume… M 28 0 0 5
15 V4 nume… <NA> 2 0 0 1
# ℹ 1 more variable: unique_rate <dbl>3.5 Sintaxis para el diagnóstico: variables numéricas
Sólo analizará las variables numéricas.
- diagnose_numeric(dataset)
- diagose_numeric(dataset, variable1, variable2, variable3, …)
diagnose_numeric(dfm)# A tibble: 36 × 10
variables min Q1 mean median Q3 max zero minus outlier
<chr> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <int> <int> <int>
1 N 1 61.5 122 122 182. 243 0 0 0
2 Edad 18 19 20.8 20 22 30 0 0 11
3 Curso 1 1 1.73 1 2 5 0 0 31
4 V1 1 3 3.53 4 4 6 0 0 23
5 V2 1 3 3.30 3 4 6 0 0 15
6 V3 1 2 3.27 3 4 6 0 0 0
7 V4 1 3 3.89 4 5 6 0 0 0
8 V5 1 1 2.70 3 3 6 0 0 0
9 V6 1 2 3.07 3 4 6 0 0 0
10 V7 1 2 3.25 3 4 6 0 0 0
# ℹ 26 more rows3.6 Sintaxis para el diagnóstico: variables categóricas
Sólo analizará las variables categóricas
- diagnose_category(dataset)
- diagnose_category(dataset, variable1, variable2, variable3, …, top=10, type=“rank”) o alternativamente rank=n en lugar de top=n, type=“rank”
- top: número de columnas que serán analizadas (e.g. top=10 incluirá variables de la columan 1 a la 10). Por defecto top=10).
- rank: indica la variable a incluir (e.g. rank=“Estudios”)
diagnose_category(dfm, rank = c(Estudios, Sexo))# A tibble: 9 × 6
variables levels N freq ratio rank
<chr> <chr> <int> <int> <dbl> <int>
1 Estudios E 243 83 34.2 1
2 Estudios C 243 71 29.2 2
3 Estudios A 243 61 25.1 3
4 Estudios D 243 23 9.47 4
5 Estudios B 243 3 1.23 5
6 Estudios <NA> 243 2 0.823 6
7 Sexo F 243 213 87.7 1
8 Sexo M 243 28 11.5 2
9 Sexo <NA> 243 2 0.823 33.7 Sintaxis para el diagnóstico: outliers
- diagonse_outliers(dataset)
- diagnose_outliers(dataset, variable1, variable2, …)
diagnose_outlier(dfm)# A tibble: 36 × 6
variables outliers_cnt outliers_ratio outliers_mean with_mean without_mean
<chr> <int> <dbl> <dbl> <dbl> <dbl>
1 N 0 0 NaN 122 122
2 Edad 11 4.53 28.5 20.8 20.4
3 Curso 31 12.8 4.87 1.73 1.27
4 V1 23 9.47 2.30 3.53 3.66
5 V2 15 6.17 2 3.30 3.42
6 V3 0 0 NaN 3.27 3.27
7 V4 0 0 NaN 3.89 3.89
8 V5 0 0 NaN 2.70 2.70
9 V6 0 0 NaN 3.07 3.07
10 V7 0 0 NaN 3.25 3.25
# ℹ 26 more rows3.8 Sintaxis para el diagnóstico: gráfico de outliers
plot_outlier(dfm, Edad, V28) # Se incluyen solamente dos variables como ejemplo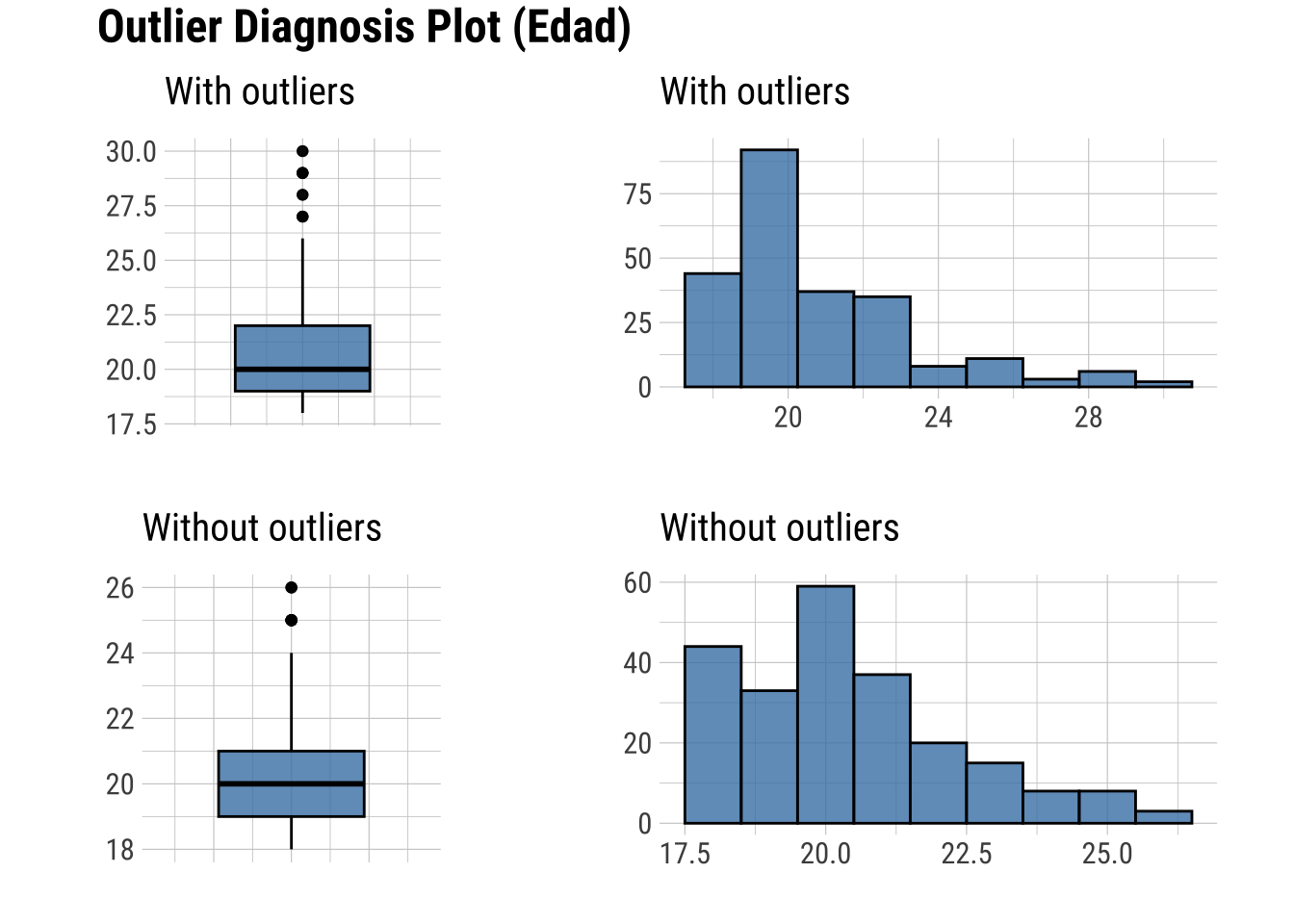
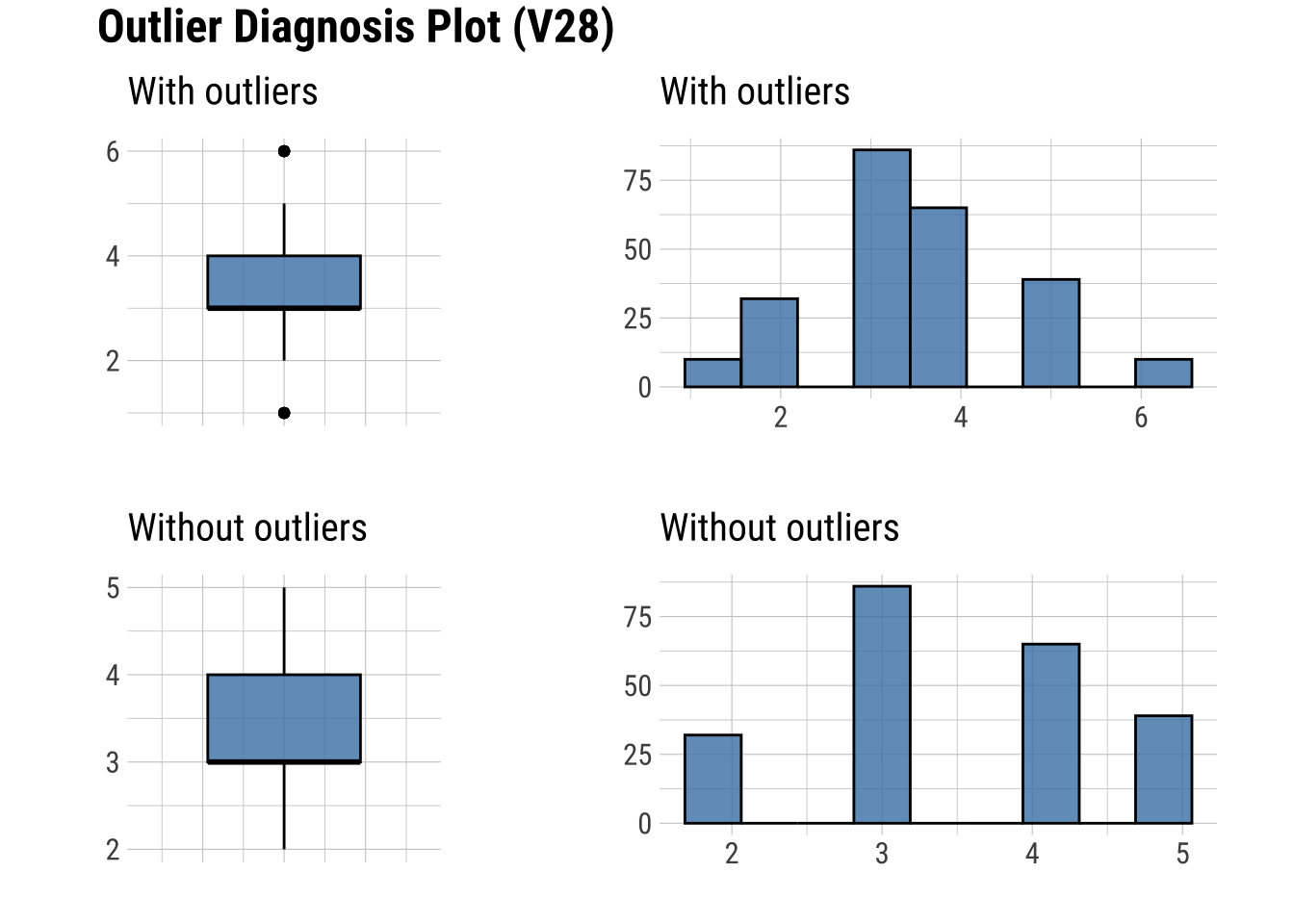
3.9 Sintaxis para el diagnóstico: datos ausentes
find_na(dfm, index = F) [1] "Sexo" "Edad" "Estudios" "V1" "V2" "V3"
[7] "V8" "V9" "V10" "V11" "V12" "V13"
[13] "V14" "V15" "V16" "V17" "V18" "V19"
[19] "V20" "V21" "V22" "V23" "V24" "V25"
[25] "V26" "V27" "V28" find_na(dfm, index = F, rate = T) # index=FALSE escribe las variables con NA's; rate=TRUE indica proporción de NA's N Universidad Sexo Edad Estudios Curso
0.000 0.000 0.823 2.058 0.823 0.000
V1 V2 V3 V4 V5 V6
2.469 23.457 13.992 0.000 0.000 0.000
V7 V8 V9 V10 V11 V12
0.000 0.412 0.412 0.823 1.235 2.058
V13 V14 V15 V16 V17 V18
2.058 2.469 2.469 2.469 2.469 2.058
V19 V20 V21 V22 V23 V24
2.058 1.646 1.646 0.823 0.823 0.823
V25 V26 V27 V28 V29 V30
0.412 0.412 0.412 0.412 0.000 0.000
V31 V32 V33
0.000 0.000 0.000 3.10 Sintaxis para el diagnóstico: datos ausentes usando dplyr
diagnose(dfm) |>
filter(missing_count > 0)# A tibble: 27 × 6
variables types missing_count missing_percent unique_count unique_rate
<chr> <chr> <int> <dbl> <int> <dbl>
1 Sexo character 2 0.823 3 0.0123
2 Edad numeric 5 2.06 14 0.0576
3 Estudios character 2 0.823 6 0.0247
4 V1 numeric 6 2.47 7 0.0288
5 V2 numeric 57 23.5 7 0.0288
6 V3 numeric 34 14.0 7 0.0288
7 V8 numeric 1 0.412 7 0.0288
8 V9 numeric 1 0.412 7 0.0288
9 V10 numeric 2 0.823 7 0.0288
10 V11 numeric 3 1.23 7 0.0288
# ℹ 17 more rows3.11 Sintaxis para el diagnóstico: gráfico de intersección de ausentes
plot_na_intersect(dfm) # : visualiza las combinaciones de valores faltantes entre columnas.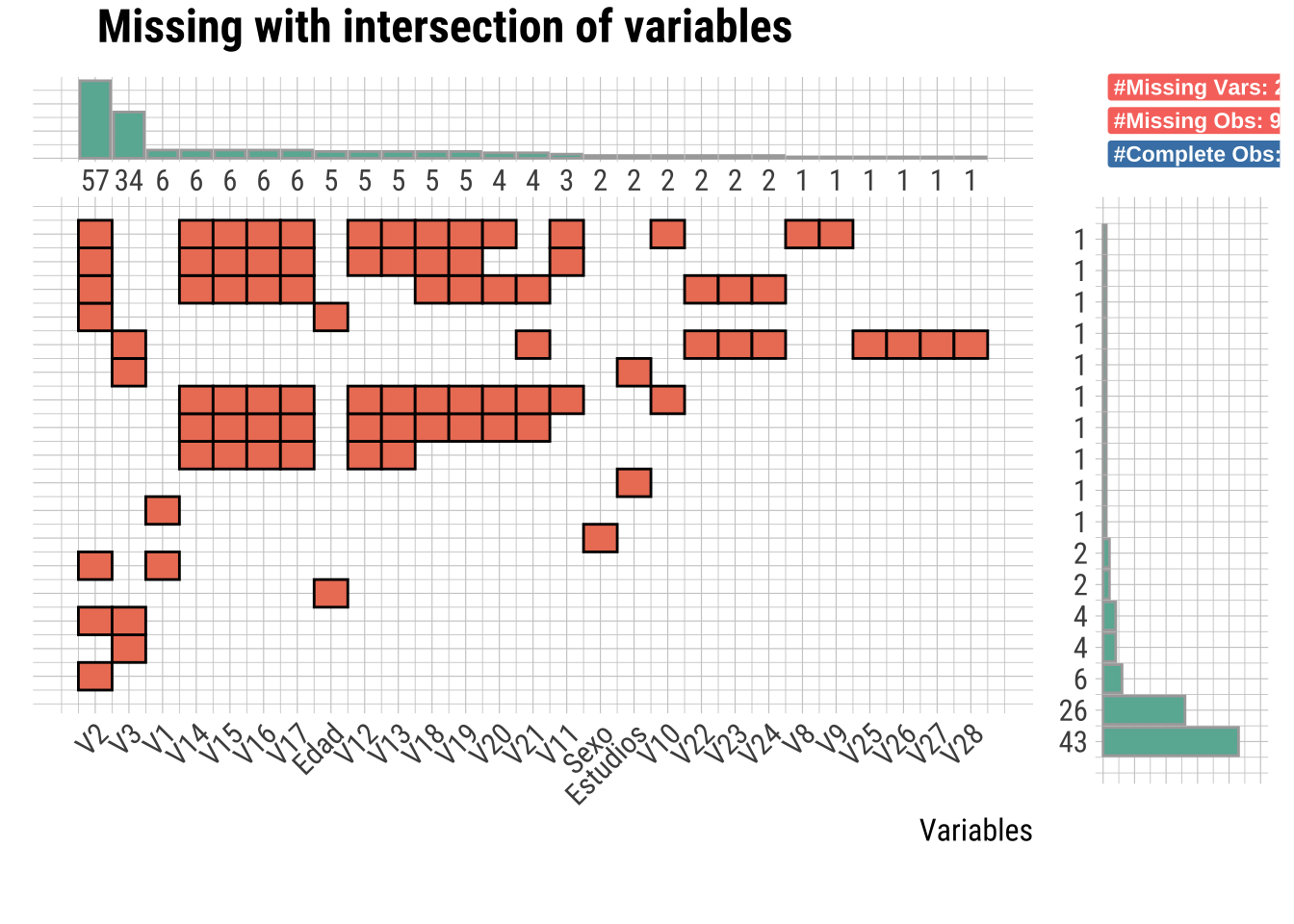
plot_na_pareto(dfm, only_na = T, plot = T) # Si plot=F obtenemos la tabla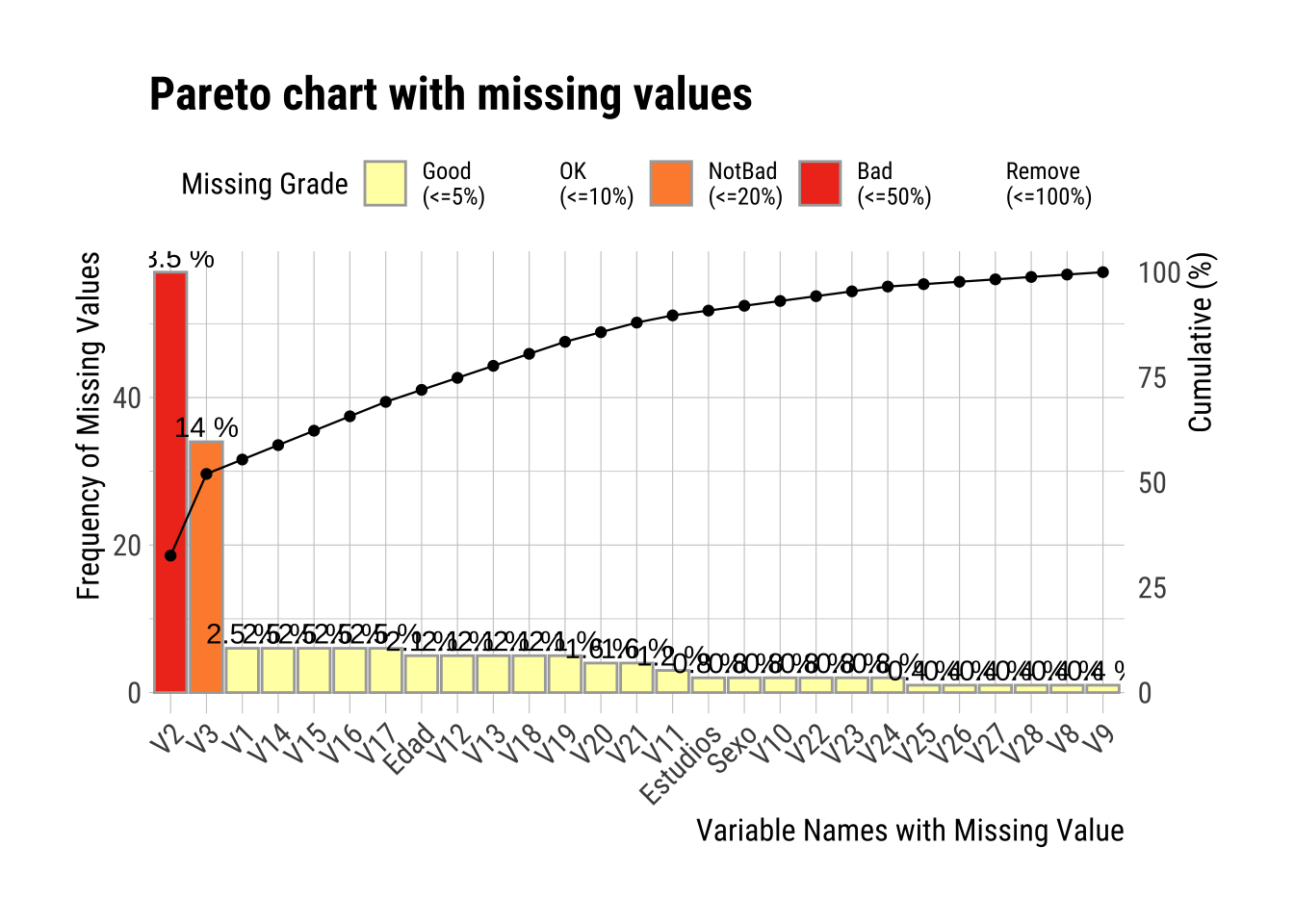
3.12 Sintaxis para tener salidas más agradables
diagnose(dfm, 2:10) %>% flextable() # Como ejemplo, sólo se han hecho con las variables desde la columna 2 a la 10variables | types | missing_count | missing_percent | unique_count | unique_rate |
|---|---|---|---|---|---|
Universidad | character | 0 | 0.0000000 | 3 | 0.01234568 |
Sexo | character | 2 | 0.8230453 | 3 | 0.01234568 |
Edad | numeric | 5 | 2.0576132 | 14 | 0.05761317 |
Estudios | character | 2 | 0.8230453 | 6 | 0.02469136 |
Curso | numeric | 0 | 0.0000000 | 5 | 0.02057613 |
V1 | numeric | 6 | 2.4691358 | 7 | 0.02880658 |
V2 | numeric | 57 | 23.4567901 | 7 | 0.02880658 |
V3 | numeric | 34 | 13.9917695 | 7 | 0.02880658 |
V4 | numeric | 0 | 0.0000000 | 6 | 0.02469136 |
4 Reparar
Al reparar, principalmente trataremos de solucionar lo siguiente:
- Los datos ausentes (NA).
- Los datos extremos (outliers).
Las dos estrategias básicas son:
- Eliminación de casos (a veces la consideración de no tener en cuenta la variable).
- Imputación de valores.
También veremos como agrupar valores (binning).
4.1 Casos NA
4.1.1 Casos con problemas en el dataset ejemplo
En el diagnóstico anterior, las variables con un elevado número de NA’s eran las siguientes:
- Edad: 2.058
- V1: 2.469
- V2: 23.457
- V3: 13.992
- V13: 2.058
- V14: 2.469
- V15: 2.469
- V16: 2.469
- V17: 2.469
- V18: 2.058
- V19: 2.058
4.1.2 Gestionar NA’s: eliminar
El paquete dlookr no tiene una función definida para eliminar casos. Se puede recurrir a otros paquetes (e.g. dplyr).
dfm_sin_na <- dfm %>%
filter(complete.cases(.))
nrow(dfm) - nrow(dfm_sin_na) # Comprobamos cuantos casos se han eliminado4.1.3 Gestionar NA’s en el dataset de ejemplo: eliminación
Eliminación casos
- Los casos con más de 5 NA’s los eliminaremos porque sospechamos que son sujetos que no son “de fiar”.
- También se quitarán los casos sin el Sexo declarado por las implicaciones que puede llevar a la hora de analizar los datos. Esto depende del objetivo/s de investigación.
# Contar los NA por fila y eliminar las que tienen 5 o más NA's
dfm_filtrado <- dfm %>%
filter(!is.na(Sexo)) %>%
mutate(na_count = rowSums(is.na(.))) %>%
filter(na_count < 5) %>%
select(-na_count) # Eliminar la columna auxiliarBuscasmos las variables que quedan con NA’s. A las que quedan, se les realizará la imputación.
find_na(dfm_filtrado, index = F)[1] "Edad" "Estudios" "V1" "V2" "V3" diagnose(dfm_filtrado, Edad, Estudios, V1, V2, V3)# A tibble: 5 × 6
variables types missing_count missing_percent unique_count unique_rate
<chr> <chr> <int> <dbl> <int> <dbl>
1 Edad numeric 5 2.14 14 0.0598
2 Estudios character 2 0.855 6 0.0256
3 V1 numeric 6 2.56 7 0.0299
4 V2 numeric 54 23.1 7 0.0299
5 V3 numeric 33 14.1 7 0.02994.1.4 Gestionar NA’s: imputación
| Método | Tipo de variable | ¿Qué hace? | Pros | Contras |
|---|---|---|---|---|
Media (mean) |
Numérica | Sustituye los valores perdidos por la media aritmética de la variable. | - Simple y rápido - Útil si la distribución es simétrica |
- Afectado por outliers - Reduce la variabilidad del conjunto |
Mediana (median) |
Numérica | Sustituye los NA por la mediana (valor central) de la variable. | - Robusta frente a valores extremos - Ideal para datos asimétricos |
- Puede crear empates artificiales |
Moda (mode) |
Numérica o Categórica | Rellena los NA con el valor más frecuente en la variable. | - Usa el valor más común - Mantiene coherencia con los datos |
- No siempre hay una moda clara - Puede sobrerrepresentar una clase |
4.1.5 Gestionar NA’s: imputación
Se puede hacer un gráfico para ver qué procedimiento se ajusta mejor. Hay que tener en cuenta otros factores (metodológicos) en cuenta.
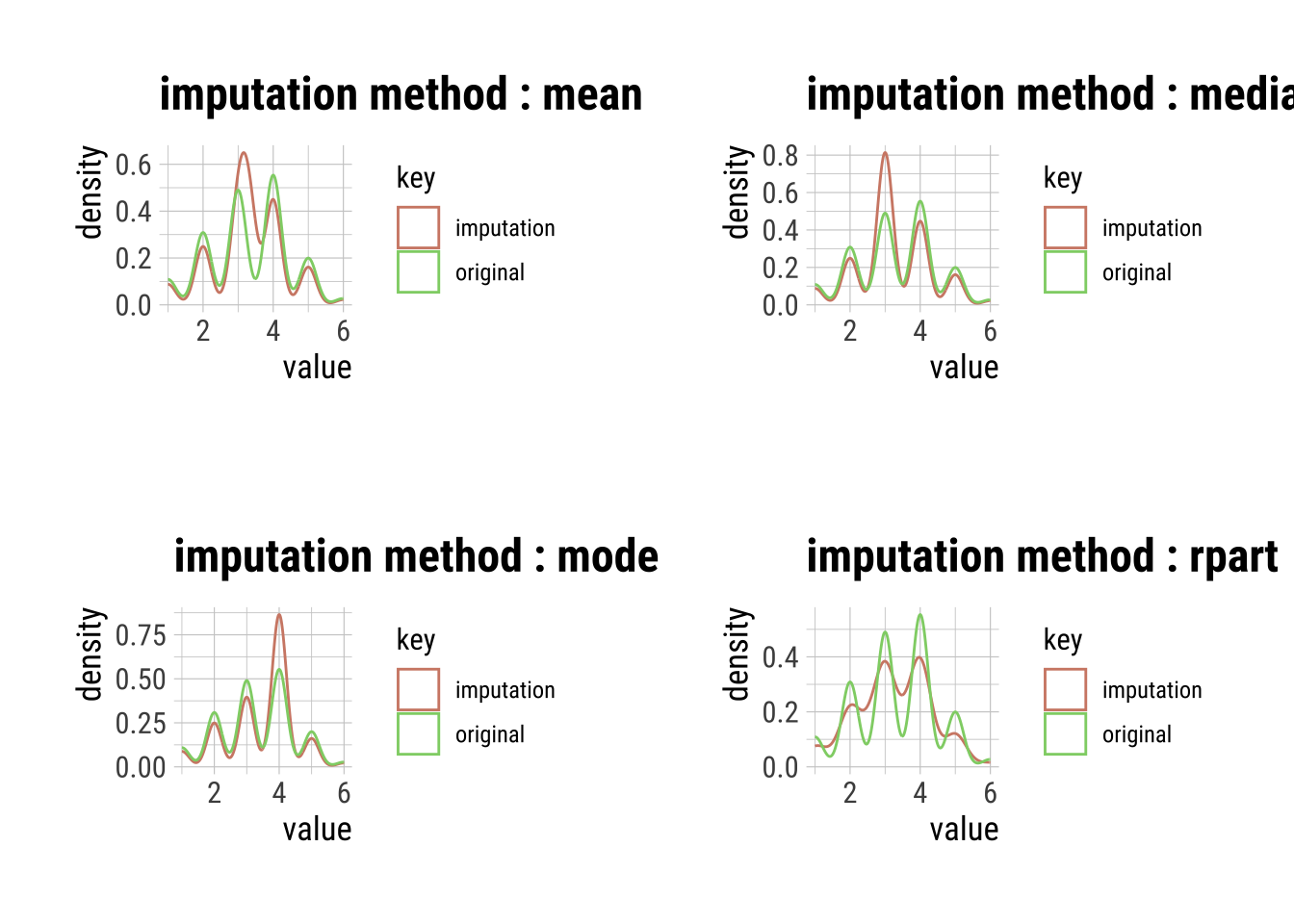
4.1.6 Gestionar NA’s en el dataset de ejemplo: imputación
Imputamos valores utilizando el método que consideramos más adecuado.
# Variable Edad (escalar)
# Media
Edad_imp <- imputate_na(dfm_filtrado, Edad, method = "mean")
summary(Edad_imp)Impute missing values with mean
* Information of Imputation (before vs after)
Original Imputation
described_variables "value" "value"
n "229" "234"
na "5" "0"
mean "20.80786" "20.80786"
sd "2.596849" "2.568834"
se_mean "0.1716046" "0.1679299"
IQR "3" "3"
skewness "1.388083" "1.402956"
kurtosis "2.009120" "2.117795"
p00 "18" "18"
p01 "18" "18"
p05 "18" "18"
p10 "18" "18"
p20 "19" "19"
p25 "19" "19"
p30 "19" "19"
p40 "20" "20"
p50 "20" "20"
p60 "21" "21"
p70 "21" "21"
p75 "22" "22"
p80 "22" "22"
p90 "24" "24"
p95 "26" "26"
p99 "29" "29"
p100 "30" "30" plot(Edad_imp)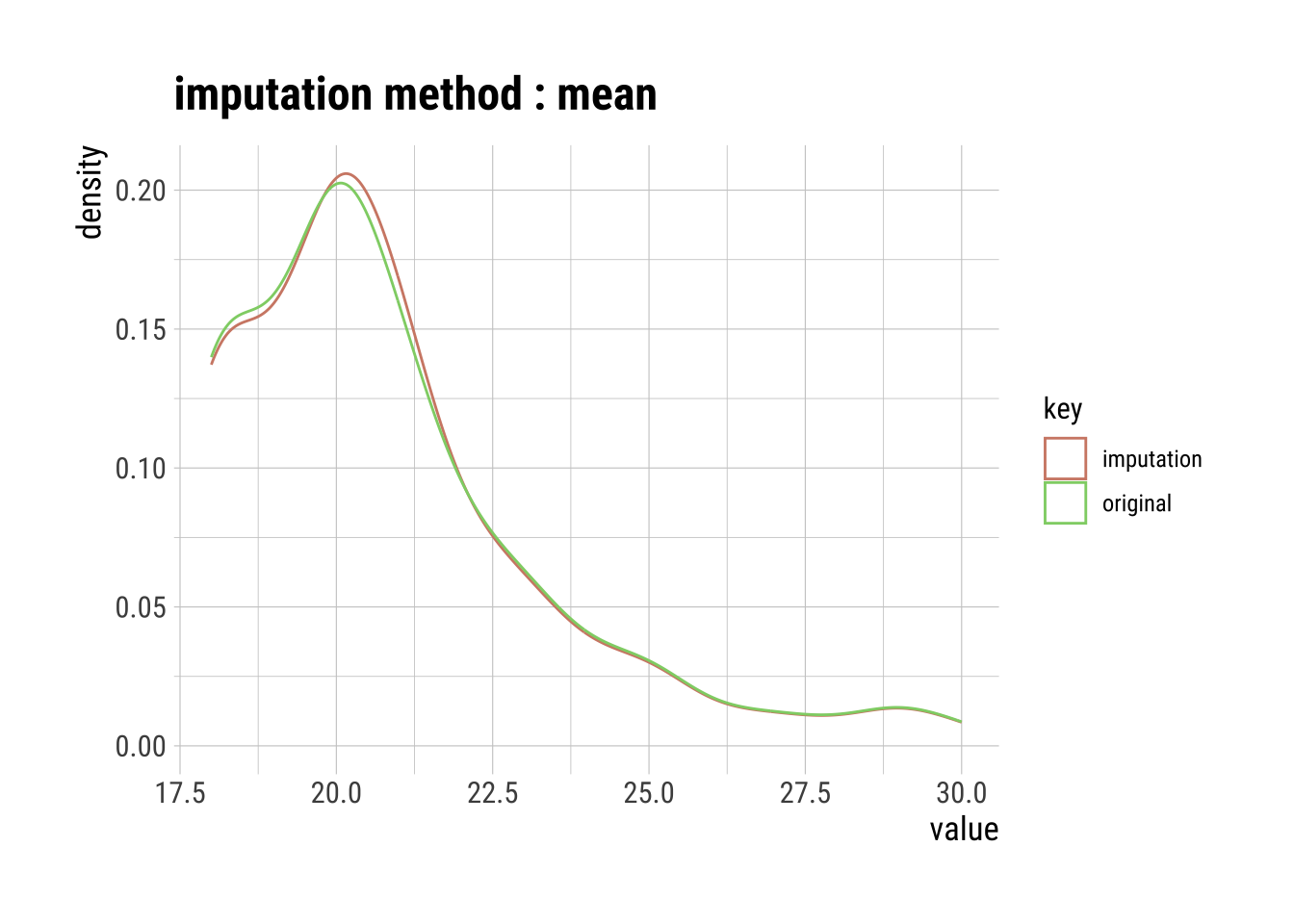
# Variable Estudios (categórica)
# Moda
## En el caso de la variable Estudios, es necesario recodificarla como números
dfm_filtrado <- dfm_filtrado %>%
mutate(Estudios_num = case_when(
Estudios == "A" ~ 1,
Estudios == "B" ~ 2,
Estudios == "C" ~ 3,
Estudios == "D" ~ 4,
Estudios == "E" ~ 5
))
Estudios_imp <- imputate_na(dfm_filtrado, Estudios_num, method = "mode")
summary(Estudios_imp)Impute missing values with mode
* Information of Imputation (before vs after)
Original Imputation
described_variables "value" "value"
n "232" "234"
na "2" "0"
mean "3.241379" "3.256410"
sd "1.552101" "1.553917"
se_mean "0.1019004" "0.1015827"
IQR "4.00" "3.75"
skewness "-0.2828929" "-0.2961816"
kurtosis "-1.324040" "-1.321206"
p00 "1" "1"
p01 "1" "1"
p05 "1" "1"
p10 "1" "1"
p20 "1" "1"
p25 "1.00" "1.25"
p30 "3" "3"
p40 "3" "3"
p50 "3" "3"
p60 "4" "4"
p70 "5" "5"
p75 "5" "5"
p80 "5" "5"
p90 "5" "5"
p95 "5" "5"
p99 "5" "5"
p100 "5" "5" plot(Estudios_imp)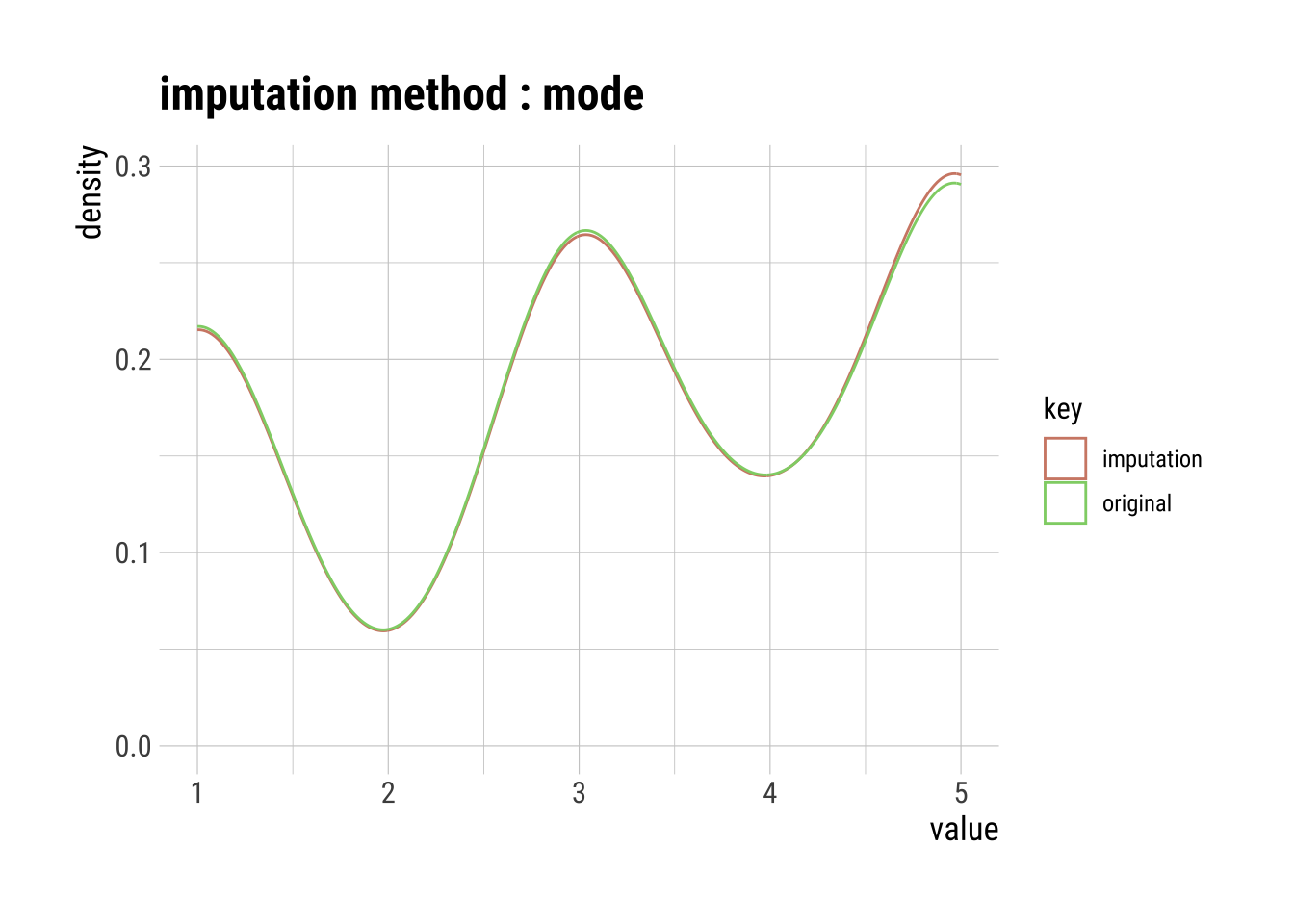
# Mediana (ordinal)
V1_imp <- imputate_na(dfm_filtrado, V1, method = "median")
summary(V1_imp)Impute missing values with median
* Information of Imputation (before vs after)
Original Imputation
described_variables "value" "value"
n "228" "234"
na "6" "0"
mean "3.517544" "3.529915"
sd "1.155202" "1.142790"
se_mean "0.07650515" "0.07470650"
IQR "1" "1"
skewness "-0.3630838" "-0.3961052"
kurtosis "-0.14065968" "-0.07398022"
p00 "1" "1"
p01 "1" "1"
p05 "1" "1"
p10 "2" "2"
p20 "3" "3"
p25 "3" "3"
p30 "3" "3"
p40 "3" "3"
p50 "4" "4"
p60 "4" "4"
p70 "4" "4"
p75 "4" "4"
p80 "4" "4"
p90 "5" "5"
p95 "5" "5"
p99 "6" "6"
p100 "6" "6" plot(V1_imp)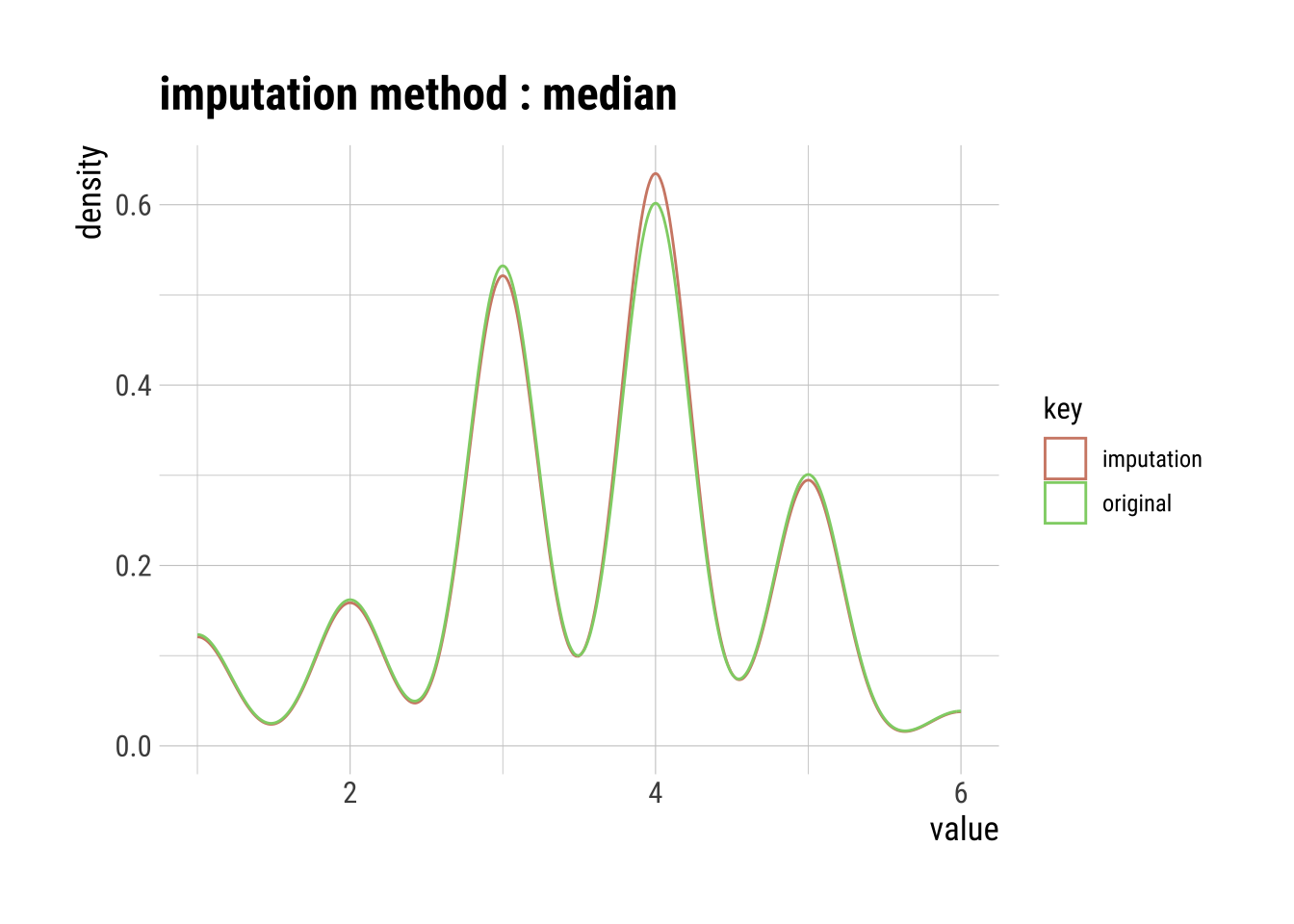
4.1.6.1 Ojo, ¿qué pasa con variables con elevadas NA’s?
Nos paramos con las variables con altos NA’s. Teniendo en cuenta que son categóricas (medida ordinal).
Variable 2
# Variable V2
# Partición recursiva y árboles de regresión
V2_imp_Mo <- imputate_na(dfm_filtrado, V2, method = "mode")
V2_imp_Md <- imputate_na(dfm_filtrado, V2, method = "median")
V2_imp_Mc <- imputate_na(dfm_filtrado, V2, method = "mice", seed = 1234)
iter imp variable
1 1 Edad V1 V2 V3 Estudios_num
1 2 Edad V1 V2 V3 Estudios_num
1 3 Edad V1 V2 V3 Estudios_num
1 4 Edad V1 V2 V3 Estudios_num
1 5 Edad V1 V2 V3 Estudios_num
2 1 Edad V1 V2 V3 Estudios_num
2 2 Edad V1 V2 V3 Estudios_num
2 3 Edad V1 V2 V3 Estudios_num
2 4 Edad V1 V2 V3 Estudios_num
2 5 Edad V1 V2 V3 Estudios_num
3 1 Edad V1 V2 V3 Estudios_num
3 2 Edad V1 V2 V3 Estudios_num
3 3 Edad V1 V2 V3 Estudios_num
3 4 Edad V1 V2 V3 Estudios_num
3 5 Edad V1 V2 V3 Estudios_num
4 1 Edad V1 V2 V3 Estudios_num
4 2 Edad V1 V2 V3 Estudios_num
4 3 Edad V1 V2 V3 Estudios_num
4 4 Edad V1 V2 V3 Estudios_num
4 5 Edad V1 V2 V3 Estudios_num
5 1 Edad V1 V2 V3 Estudios_num
5 2 Edad V1 V2 V3 Estudios_num
5 3 Edad V1 V2 V3 Estudios_num
5 4 Edad V1 V2 V3 Estudios_num
5 5 Edad V1 V2 V3 Estudios_numWarning: Number of logged events: 3V2_imp_R <- imputate_na(dfm_filtrado, V2, method = "rpart")
# par(mfrow = c(2, 2))
p1 <- plot(V2_imp_Mo)
p2 <- plot(V2_imp_Md)
p3 <- plot(V2_imp_Mc)
p4 <- plot(V2_imp_R)
grid.arrange(p1, p2, p3, p4, ncol = 2)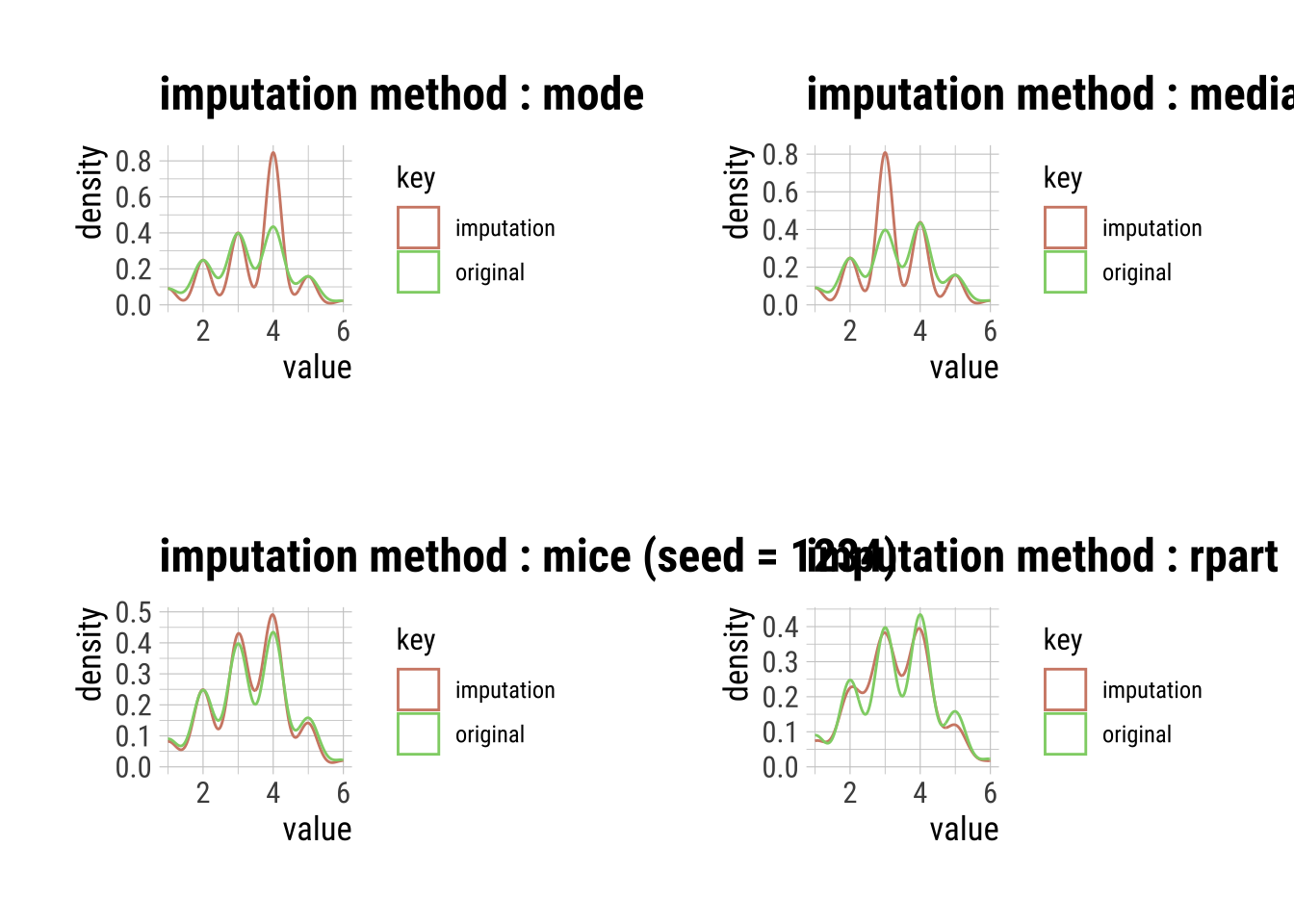
Variable 3
# Variable V3
# Partición recursiva y árboles de regresión
V3_imp_Mo <- imputate_na(dfm_filtrado, V3, method = "mode")
V3_imp_Md <- imputate_na(dfm_filtrado, V3, method = "median")
V3_imp_Mc <- imputate_na(dfm_filtrado, V3, method = "mice", seed = 1234)
iter imp variable
1 1 Edad V1 V2 V3 Estudios_num
1 2 Edad V1 V2 V3 Estudios_num
1 3 Edad V1 V2 V3 Estudios_num
1 4 Edad V1 V2 V3 Estudios_num
1 5 Edad V1 V2 V3 Estudios_num
2 1 Edad V1 V2 V3 Estudios_num
2 2 Edad V1 V2 V3 Estudios_num
2 3 Edad V1 V2 V3 Estudios_num
2 4 Edad V1 V2 V3 Estudios_num
2 5 Edad V1 V2 V3 Estudios_num
3 1 Edad V1 V2 V3 Estudios_num
3 2 Edad V1 V2 V3 Estudios_num
3 3 Edad V1 V2 V3 Estudios_num
3 4 Edad V1 V2 V3 Estudios_num
3 5 Edad V1 V2 V3 Estudios_num
4 1 Edad V1 V2 V3 Estudios_num
4 2 Edad V1 V2 V3 Estudios_num
4 3 Edad V1 V2 V3 Estudios_num
4 4 Edad V1 V2 V3 Estudios_num
4 5 Edad V1 V2 V3 Estudios_num
5 1 Edad V1 V2 V3 Estudios_num
5 2 Edad V1 V2 V3 Estudios_num
5 3 Edad V1 V2 V3 Estudios_num
5 4 Edad V1 V2 V3 Estudios_num
5 5 Edad V1 V2 V3 Estudios_numWarning: Number of logged events: 3V3_imp_R <- imputate_na(dfm_filtrado, V3, method = "rpart")
# par(mfrow = c(2, 2))
p1 <- plot(V3_imp_Mo)
p2 <- plot(V3_imp_Md)
p3 <- plot(V3_imp_Mc)
p4 <- plot(V3_imp_R)
grid.arrange(p1, p2, p3, p4, ncol = 2)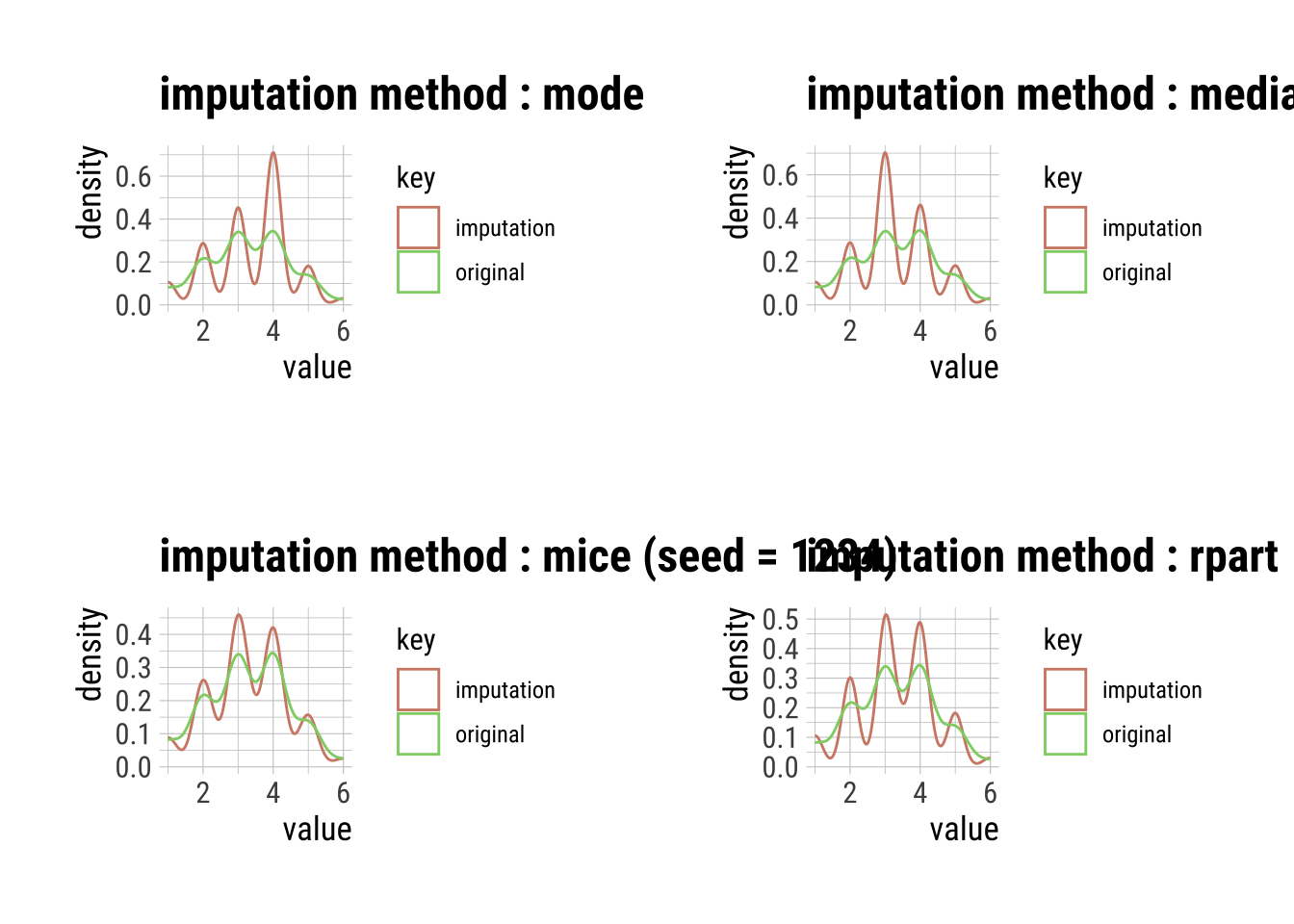
4.1.7 Crear dataset nuevo (provisional)
Una vez con las variables imputadas, nos “apañamos” un dataset temporal que recoga los casos sin NA’s que llevamos hasta el momento:
dfm_filtrado$Edad_imp <- Edad_imp
dfm_filtrado$Estudios_imp <- Estudios_imp
dfm_filtrado$V1_imp <- V1_imp
dfm_filtrado$V2_imp <- V2_imp_Mc
dfm_filtrado$V3_imp <- V3_imp_Mc
dim(dfm_filtrado)[1] 234 454.2 Outliers
4.2.1 Encontrar variables con outliers
find_outliers(dfm_filtrado, index = F) [1] "Edad" "Curso" "V1" "V8" "V14" "V16" "V18" "V19" "V20"
[10] "V22" "V25" "V28" De las que aparecen, no nos interesan Edad (porque trabajamos ya con Edad_imp), Curso (porque la muestra estaba ya sesgada desde origen), ni V1 (porque trabajamos ya con V1_imp)
Realizamos un nuevo diagnóstico para valorar qué variables se deberían transformar, en función de criterios dirigidos por el objetivo de investigación (e.g. por diferencias grandes en los promedios).
dagnos <- diagnose_outlier(dfm_filtrado, V8, V14, V16, V18, V19, V20, V22, V25, V28)
dife <- dagnos$with_mean - dagnos$without_mean
tabla <- cbind(dagnos, dife)
tabla variables outliers_cnt outliers_ratio outliers_mean with_mean without_mean
1 V8 21 8.974359 1.952381 3.354701 3.492958
2 V14 30 12.820513 2.166667 3.435897 3.622549
3 V16 25 10.683761 3.000000 3.427350 3.478469
4 V18 28 11.965812 2.964286 3.222222 3.257282
5 V19 14 5.982906 5.285714 2.790598 2.631818
6 V20 15 6.410256 5.266667 2.705128 2.529680
7 V22 14 5.982906 5.285714 2.799145 2.640909
8 V25 22 9.401709 5.500000 2.683761 2.391509
9 V28 20 8.547009 3.500000 3.529915 3.532710
dife
1 -0.13825689
2 -0.18665158
3 -0.05111847
4 -0.03505933
5 0.15878011
6 0.17544784
7 0.15823621
8 0.29225125
9 -0.00279575plot_outlier(dfm_filtrado, V8, V14, V16, V18, V19, V20, V22, V25, V28)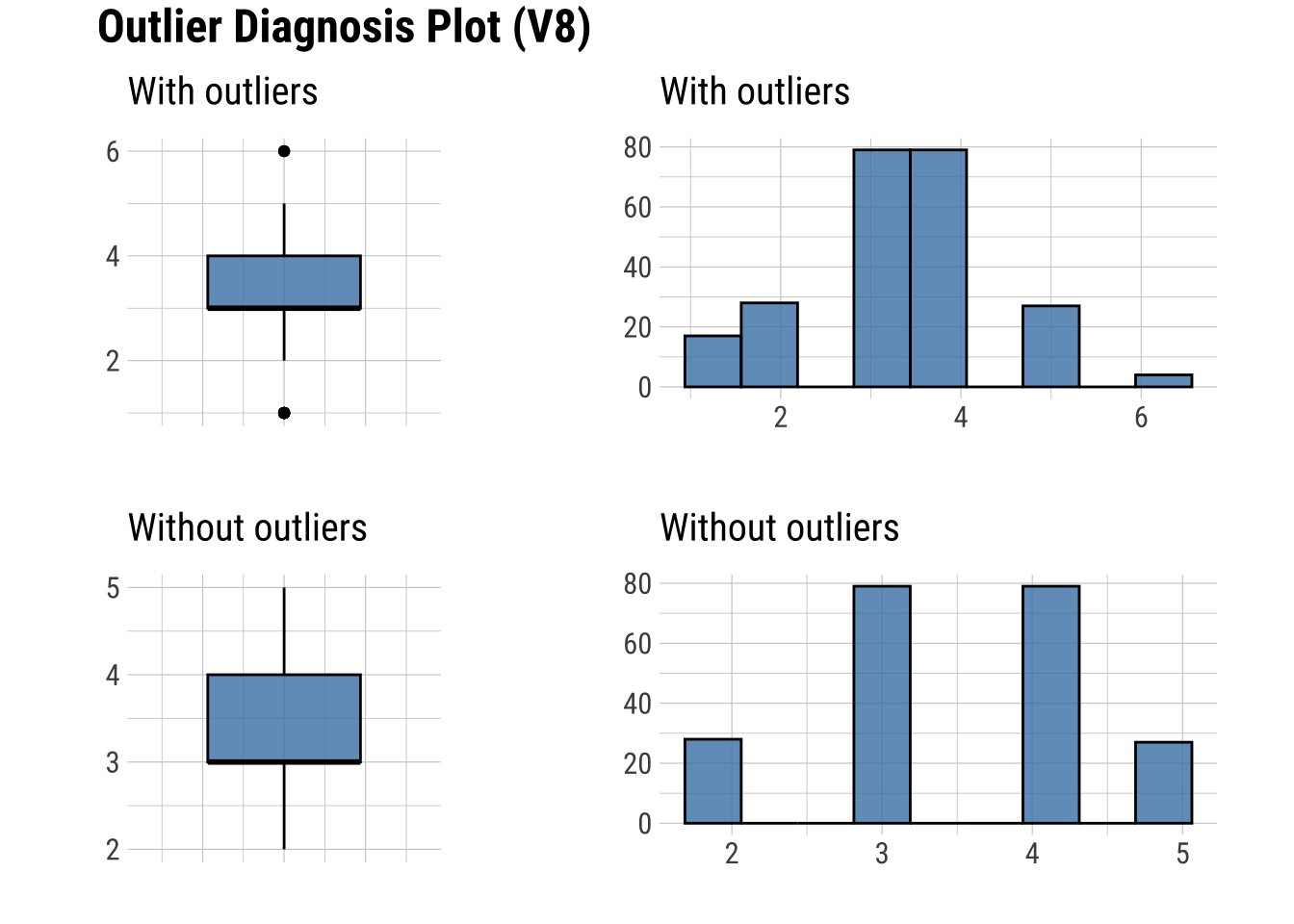
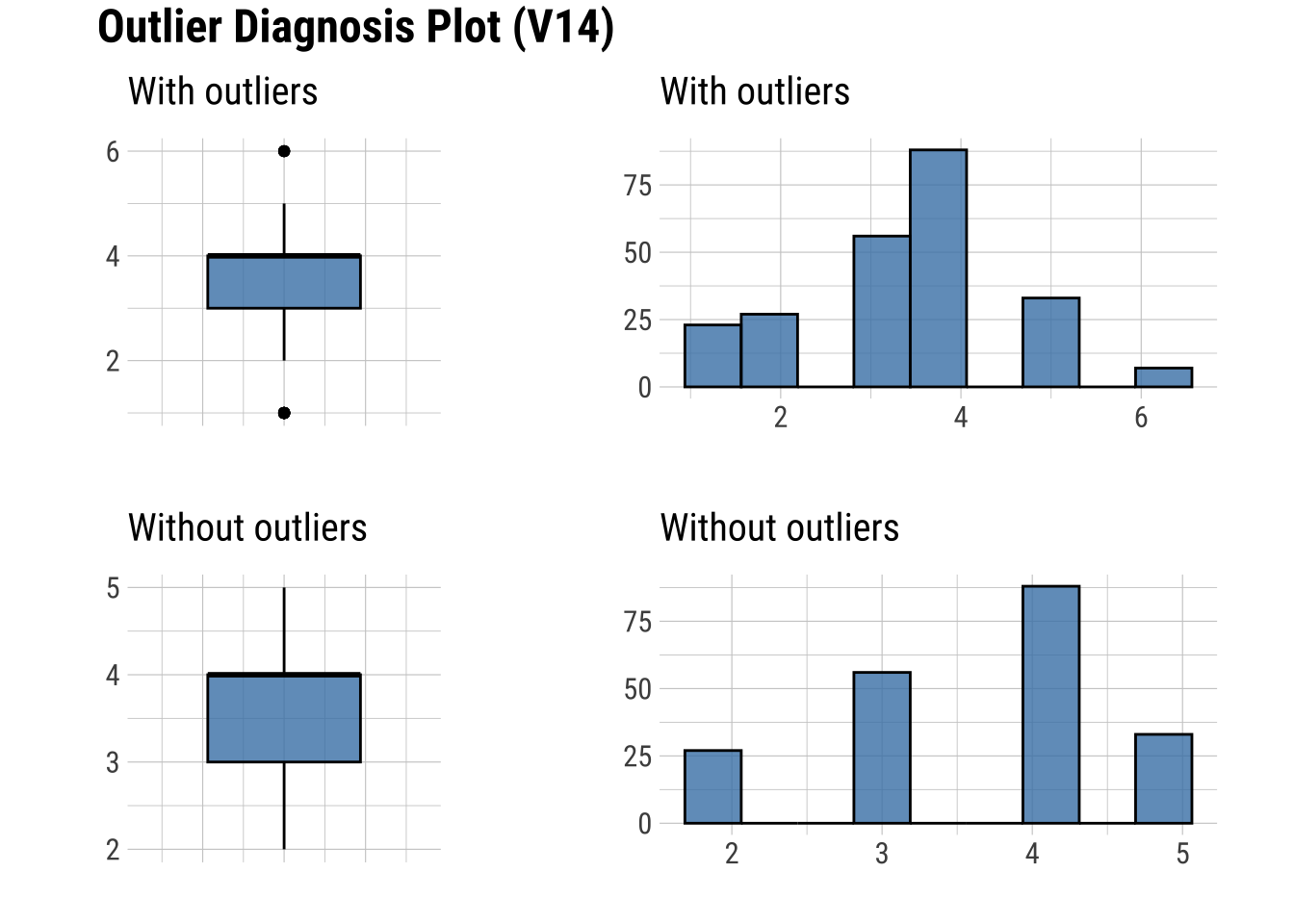
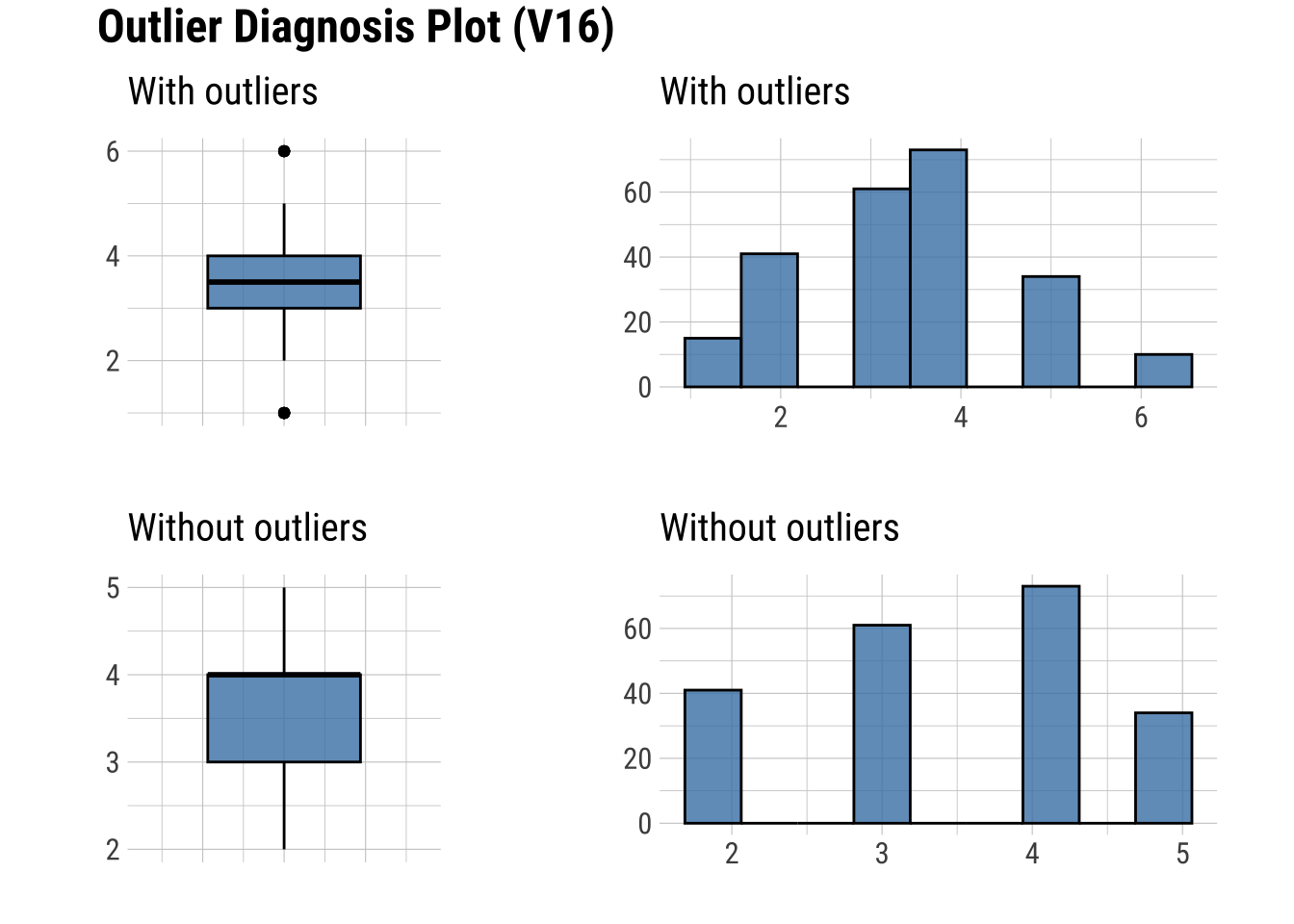
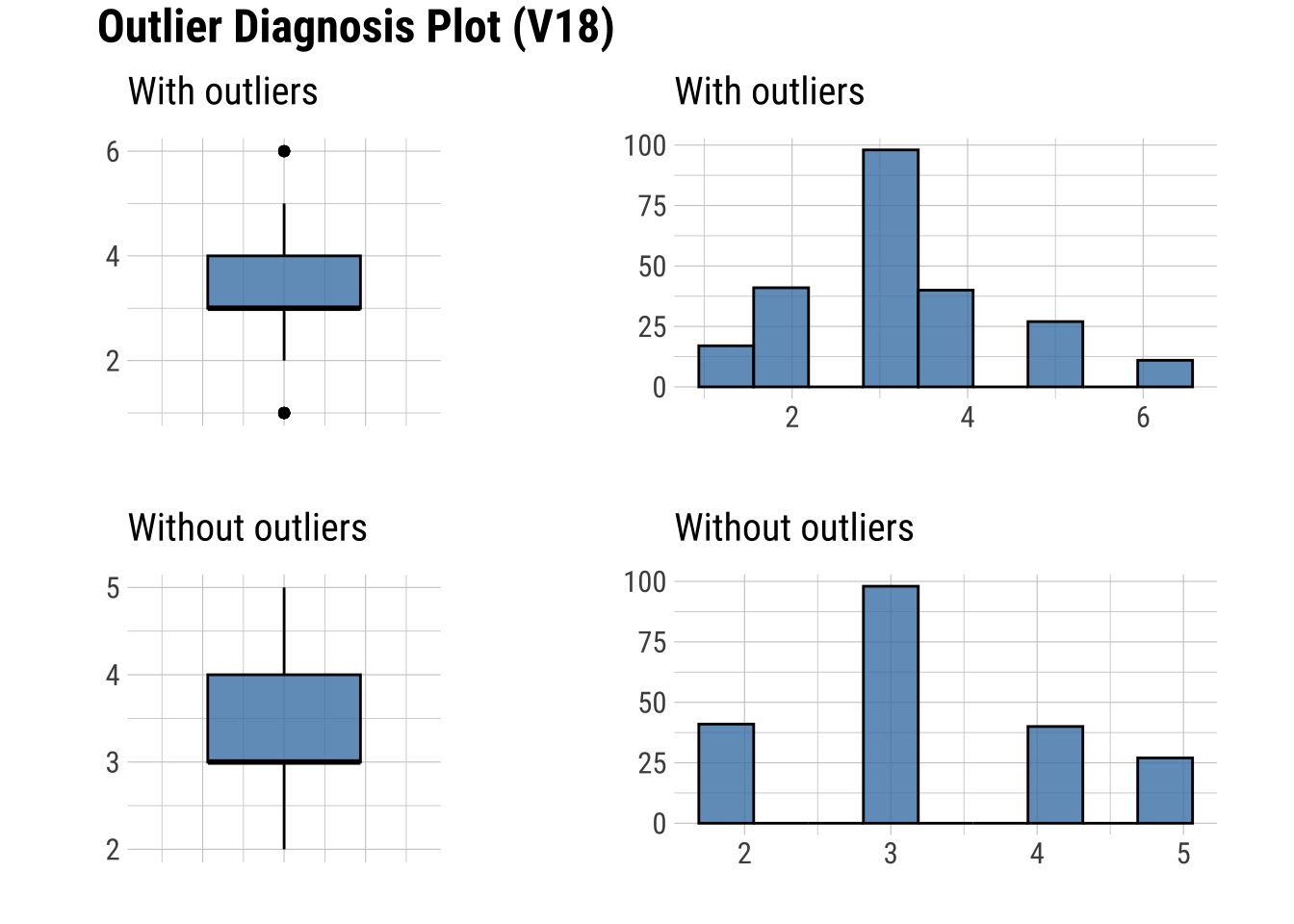
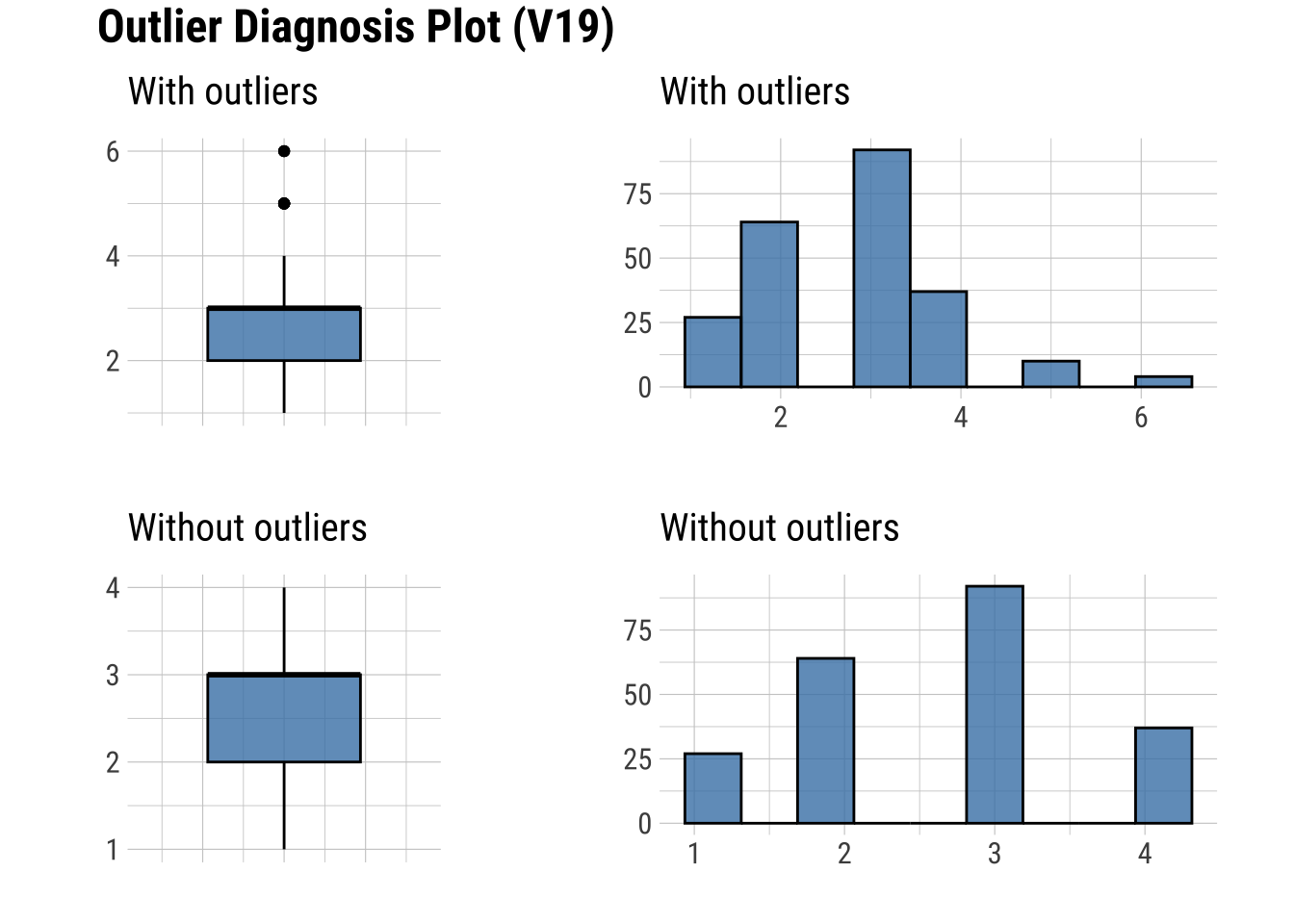
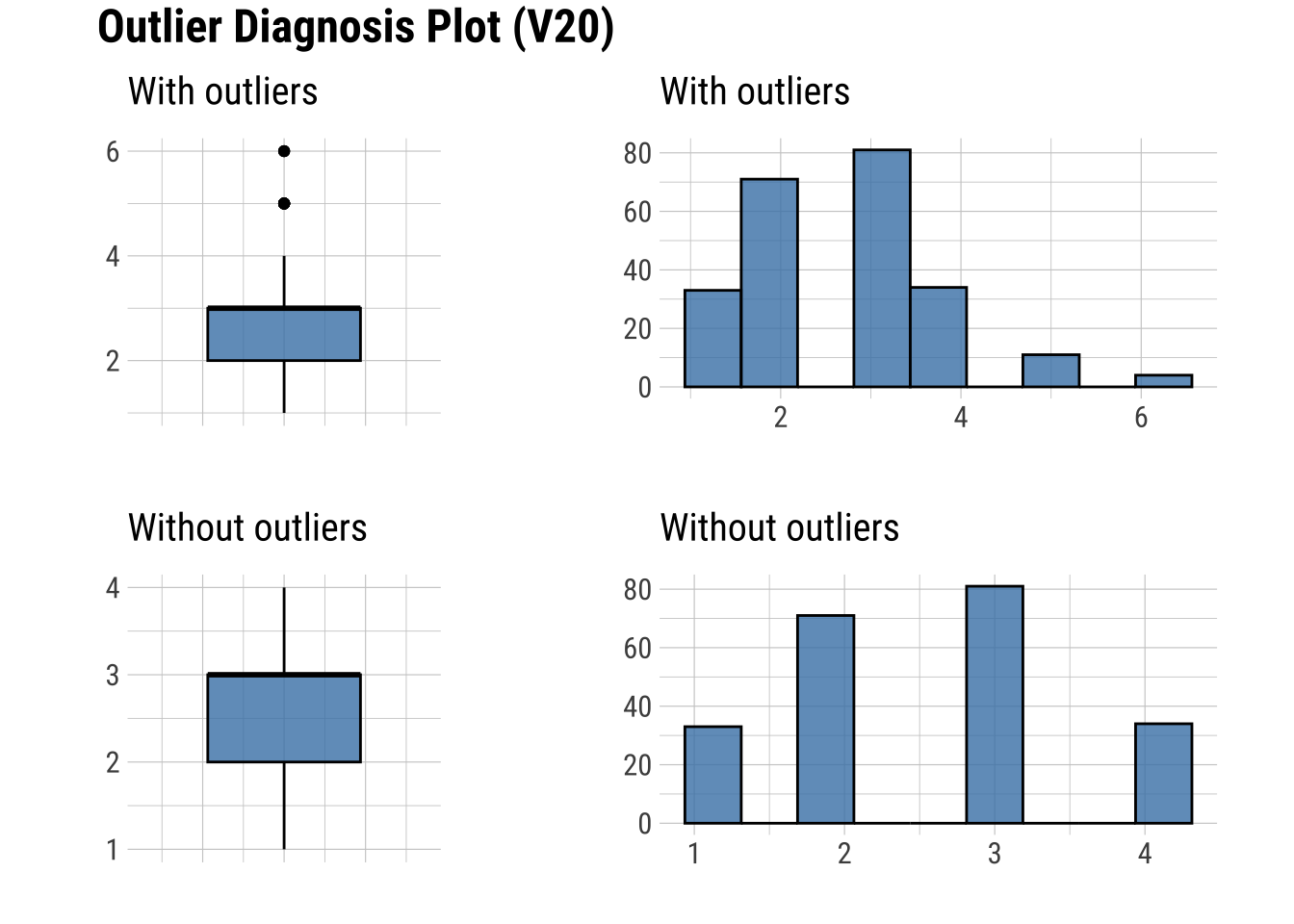
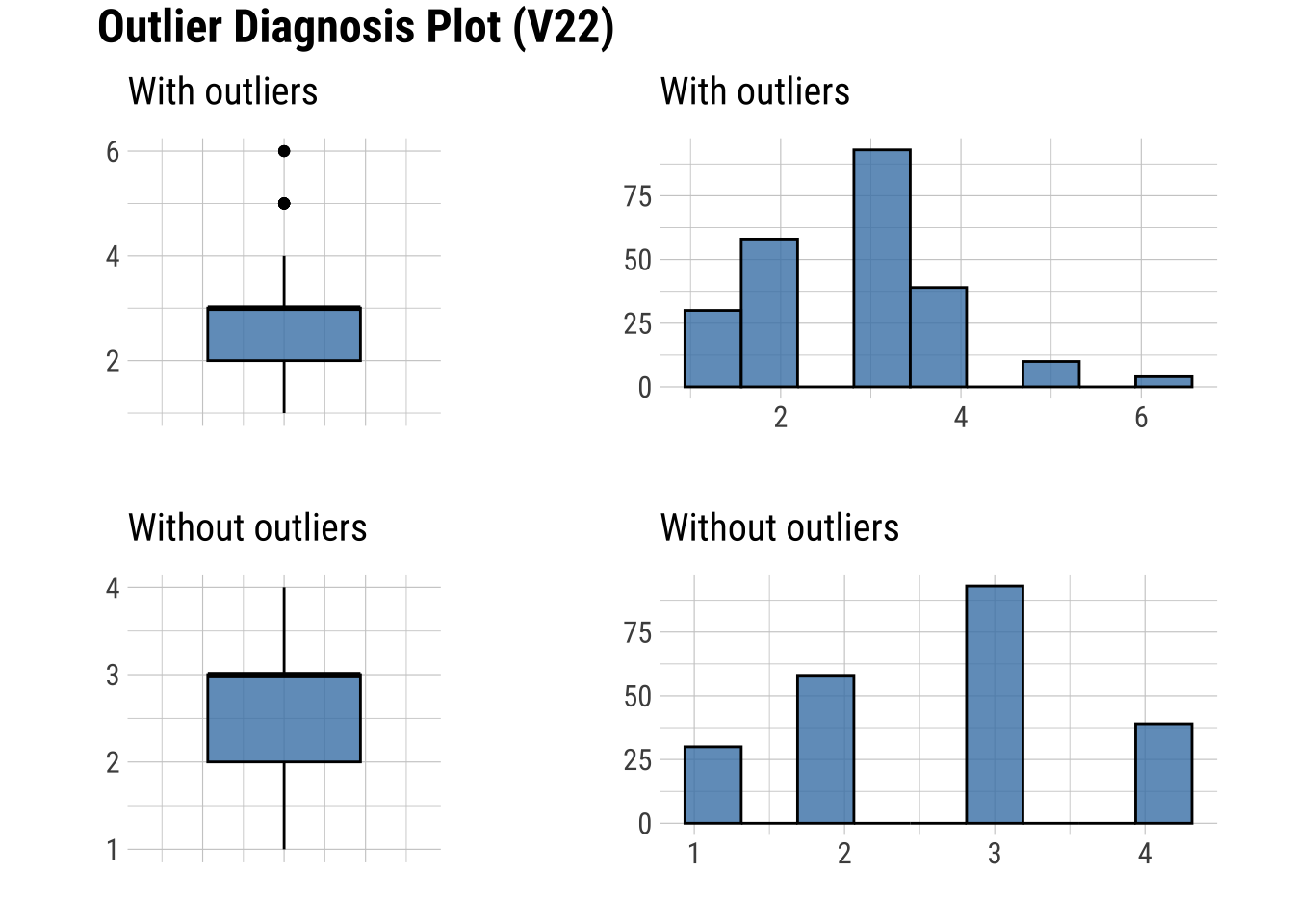
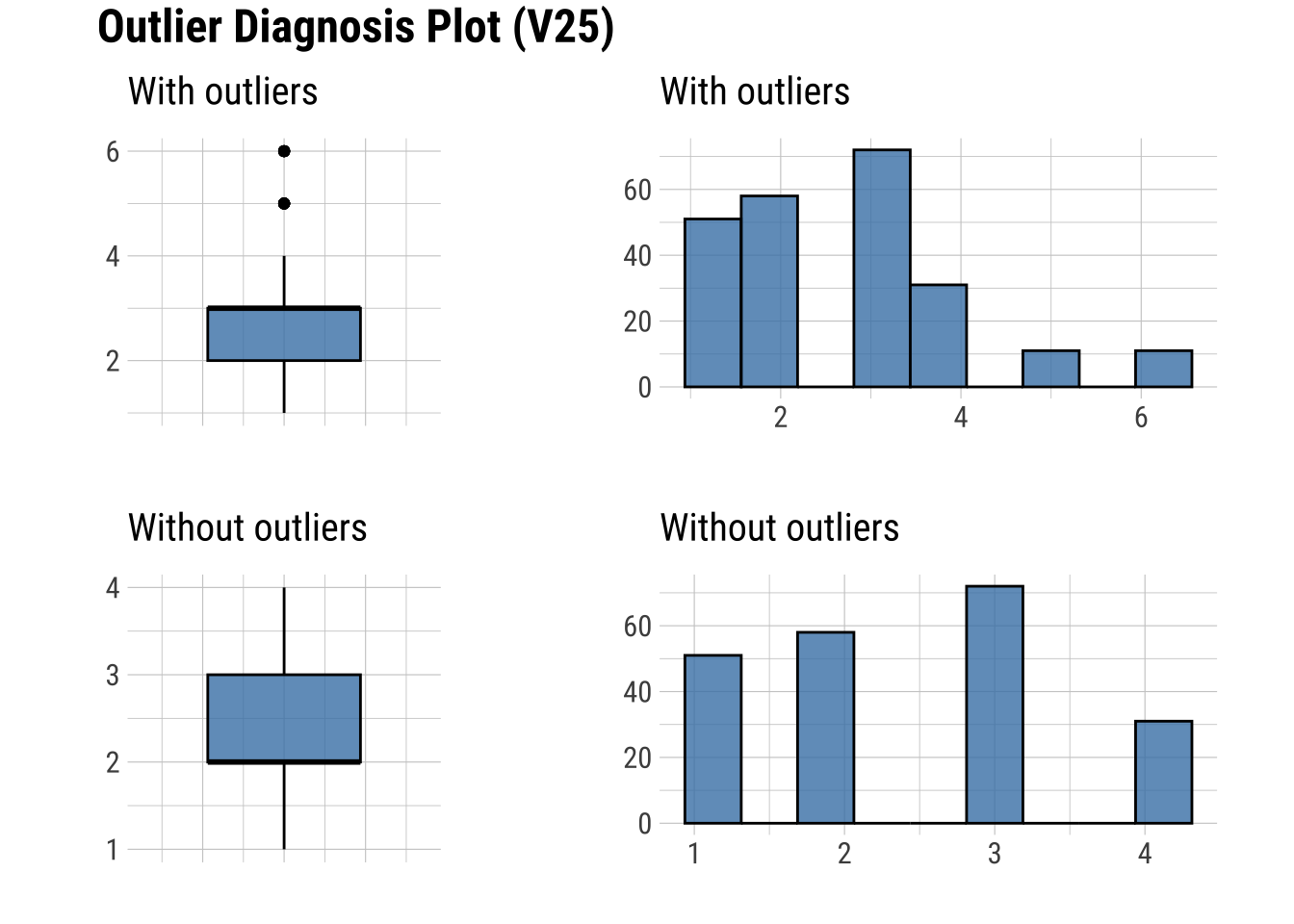
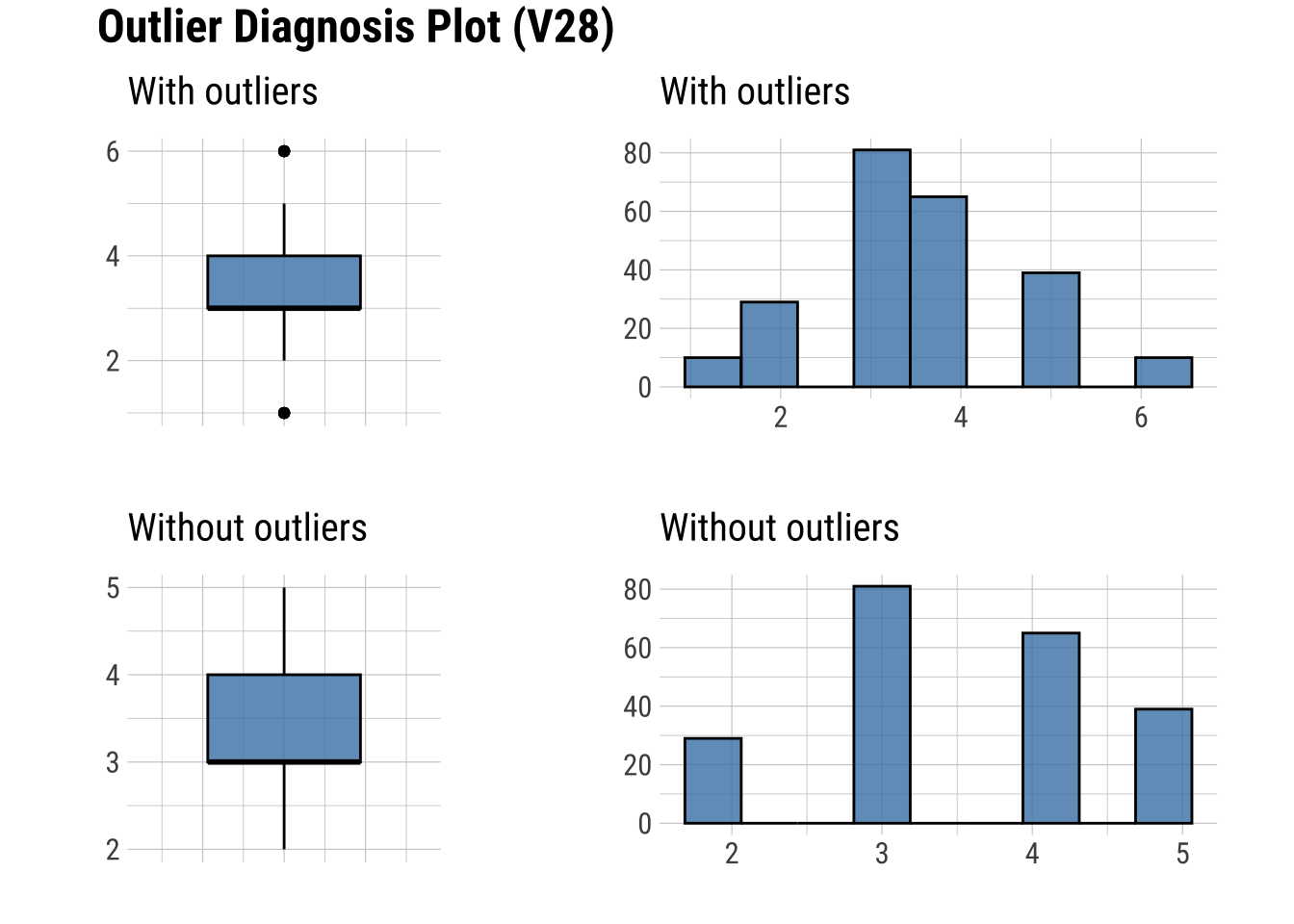
Se observan cambios destacados en V25.
4.2.2 Gestionar outliers: eliminar casos
- Una posibilidad es eliminarlos.
- Se va a realizar sólo como ejemplo,
- sin embargo, en los datos finales no se realizará porque el % de eliminación alcanzaría casi el 10% de los casos (ver tabla de diagnóstico de outliers)
Lo hacemos con la variable V8 como ejemplo.
Q1 <- quantile(dfm_filtrado$V8, .25)
Q3 <- quantile(dfm_filtrado$V8, .75)
IQR <- IQR(dfm_filtrado$V8)
no_outliers <- subset(dfm_filtrado, dfm_filtrado$V8 > (Q1 - 1.5 * IQR) & dfm_filtrado$V8 < (Q3 + 1.5 * IQR))
NROW(no_outliers)[1] 2134.2.3 Gestionar outliers: imputación
Varible V25
# Outliers V25:
p1 <- plot(imputate_outlier(dfm_filtrado, V25, method = "mean"))
p2 <- plot(imputate_outlier(dfm_filtrado, V25, method = "median"))
p3 <- plot(imputate_outlier(dfm_filtrado, V25, method = "mode"))
p4 <- plot(imputate_outlier(dfm_filtrado, V25, method = "capping"))
grid.arrange(p1, p2, p3, p4, ncol = 2)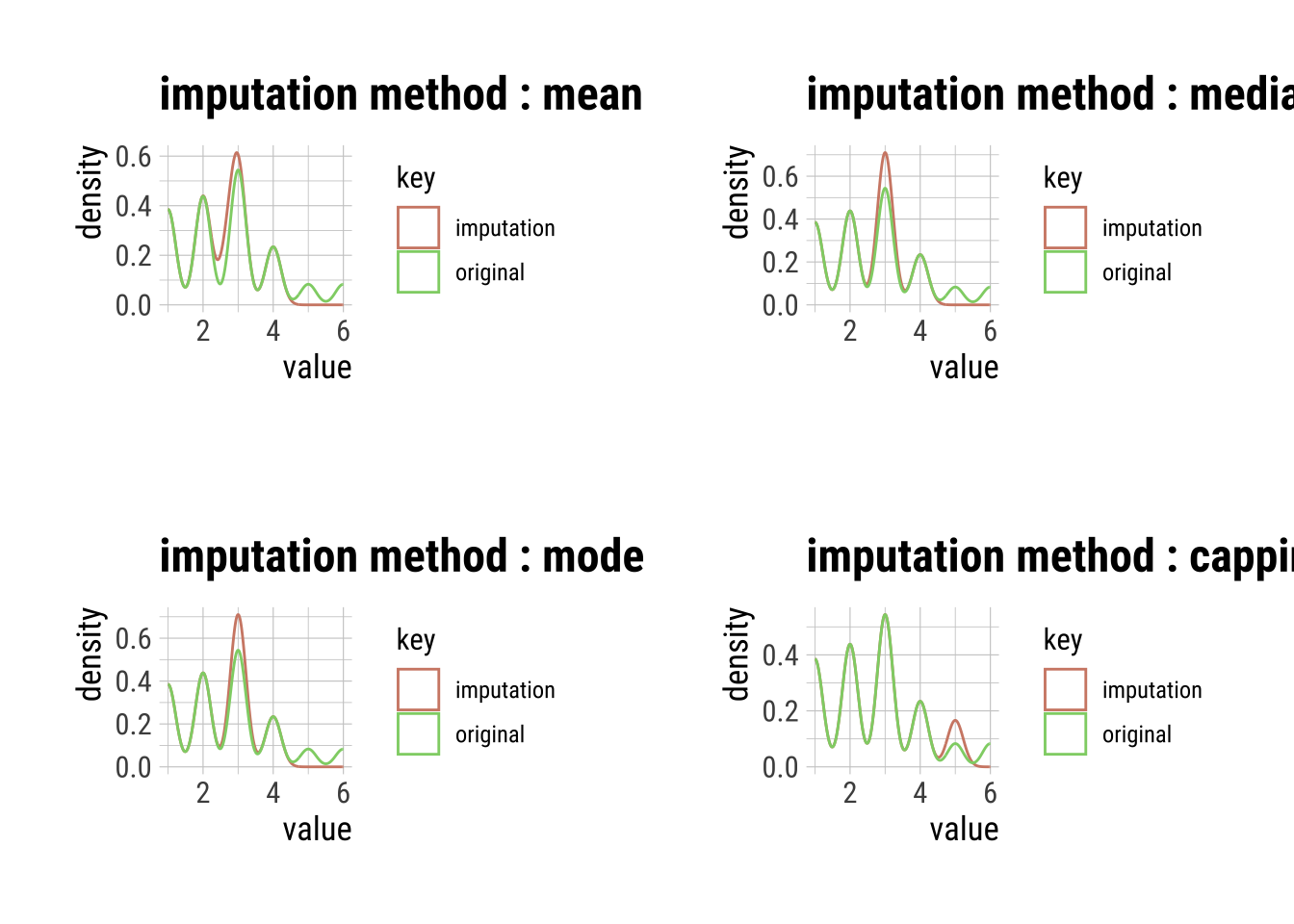
# Nos quedamos con la transformación más respetuosa
V25_imp <- imputate_outlier(dfm_filtrado, V25, method = "capping")
dfm_filtrado$V25_imp <- V25_imp4.3 Agrupamiento (binning):
Dlookr incluye los siguientes procedimientos:
- quantil (segmentación por cuartiles, usado por defecto),
- equal (segmentos igual tamaño),
- pretty (elige tamaños buenos-bonitos ¿?),
- Otros como k-means y bclust (vía bagging y bootstrap aggregating).
4.4 Agrupamiento de la variable Edad del dataset de ejemplo
# Vemos que agrupación es más útil.
plot(binning(dfm_filtrado$Edad_imp, type = "quantile", nbins = 4))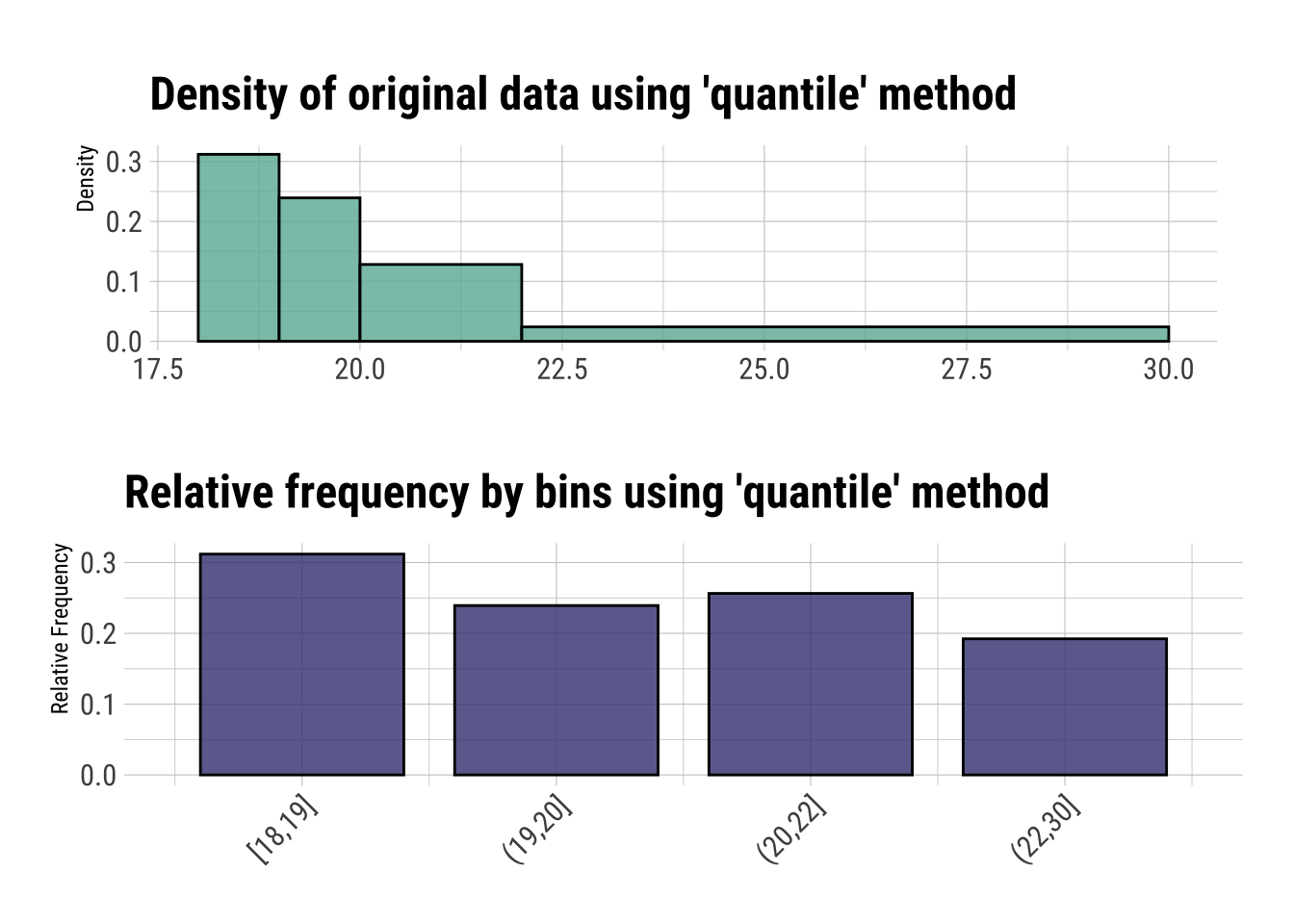
plot(binning(dfm_filtrado$Edad_imp, type = "equal", nbins = 4))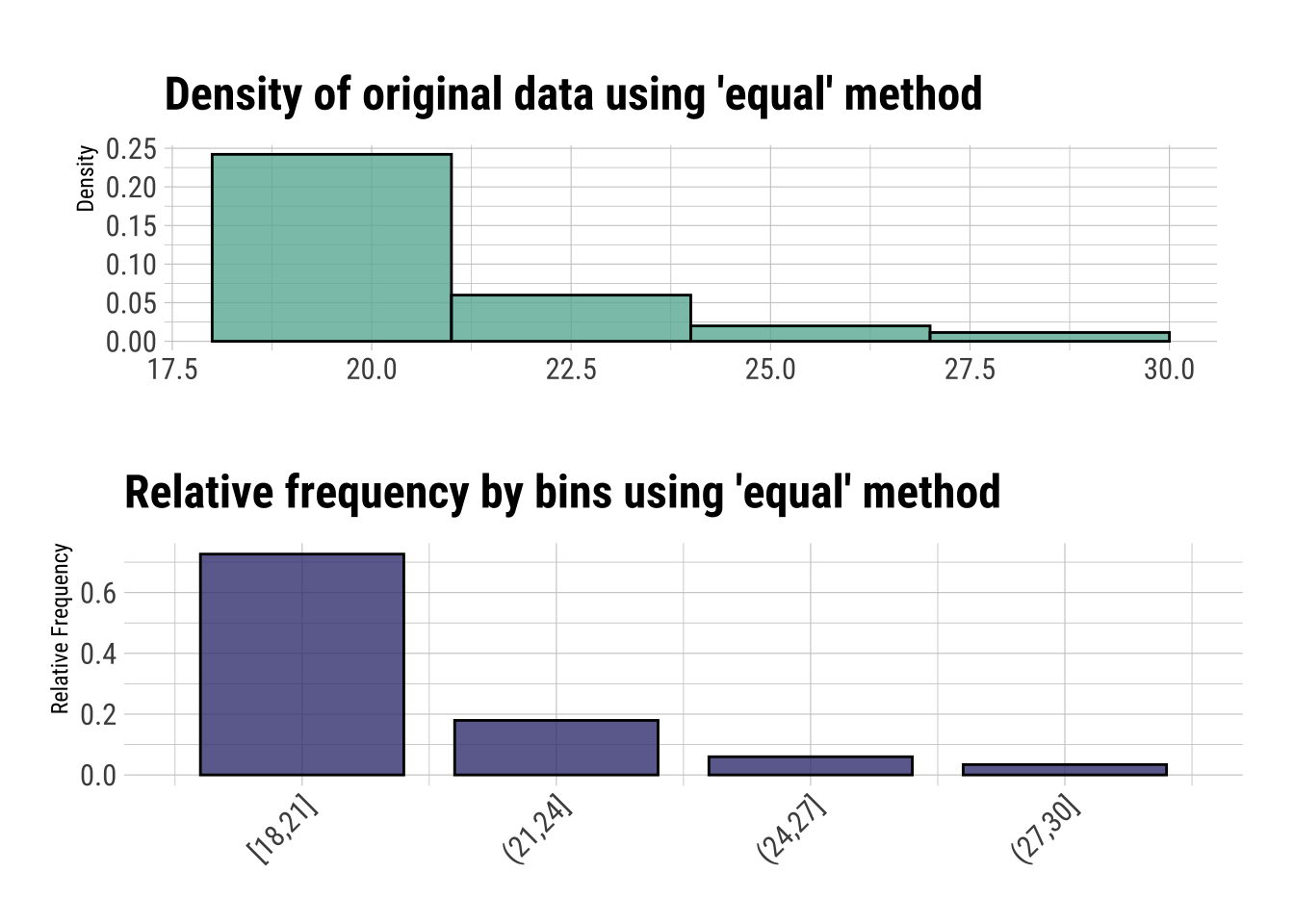
plot(binning(dfm_filtrado$Edad_imp, type = "pretty", nbins = 4))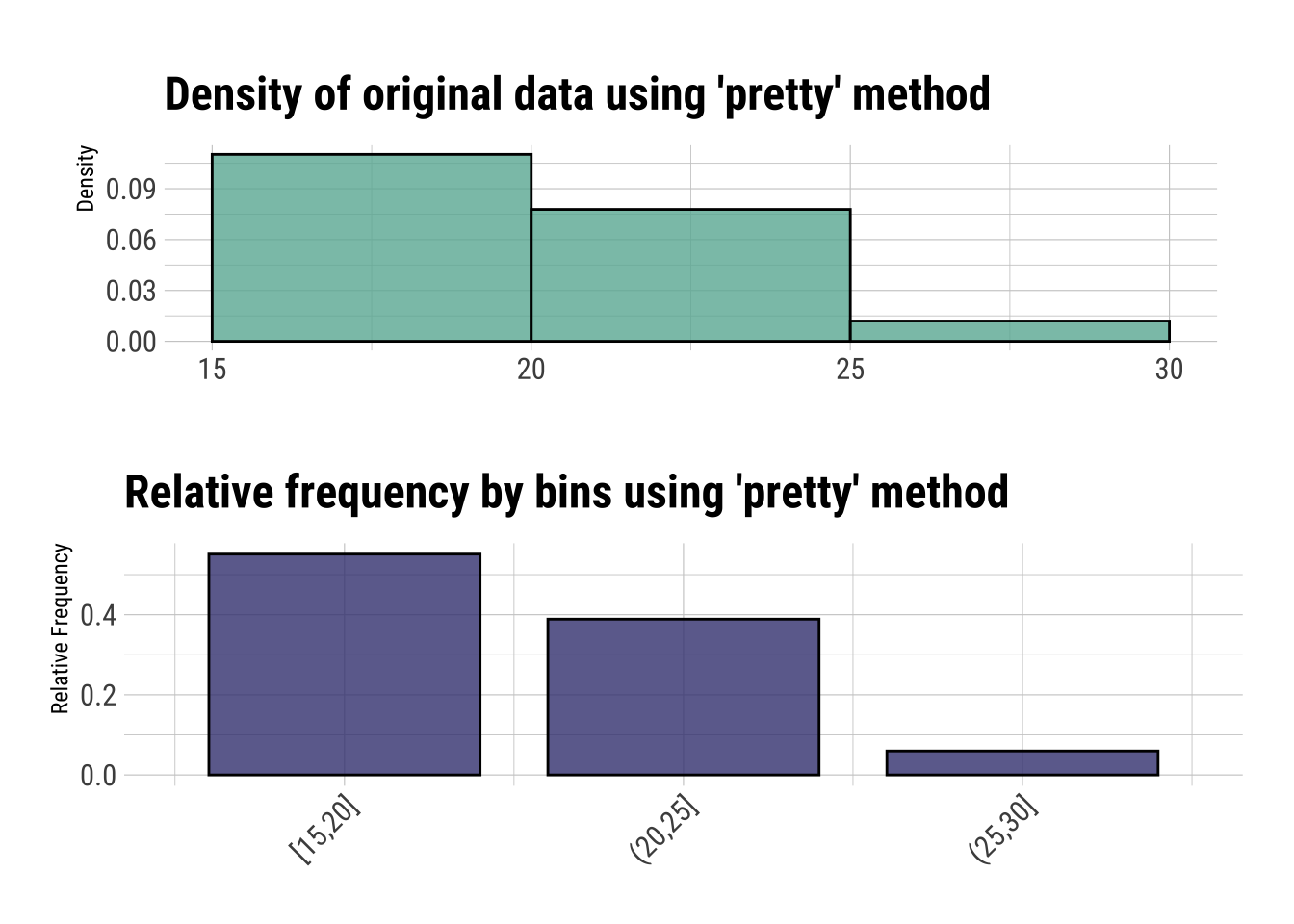
plot(binning(dfm_filtrado$Edad_imp, type = "kmeans", nbins = 4))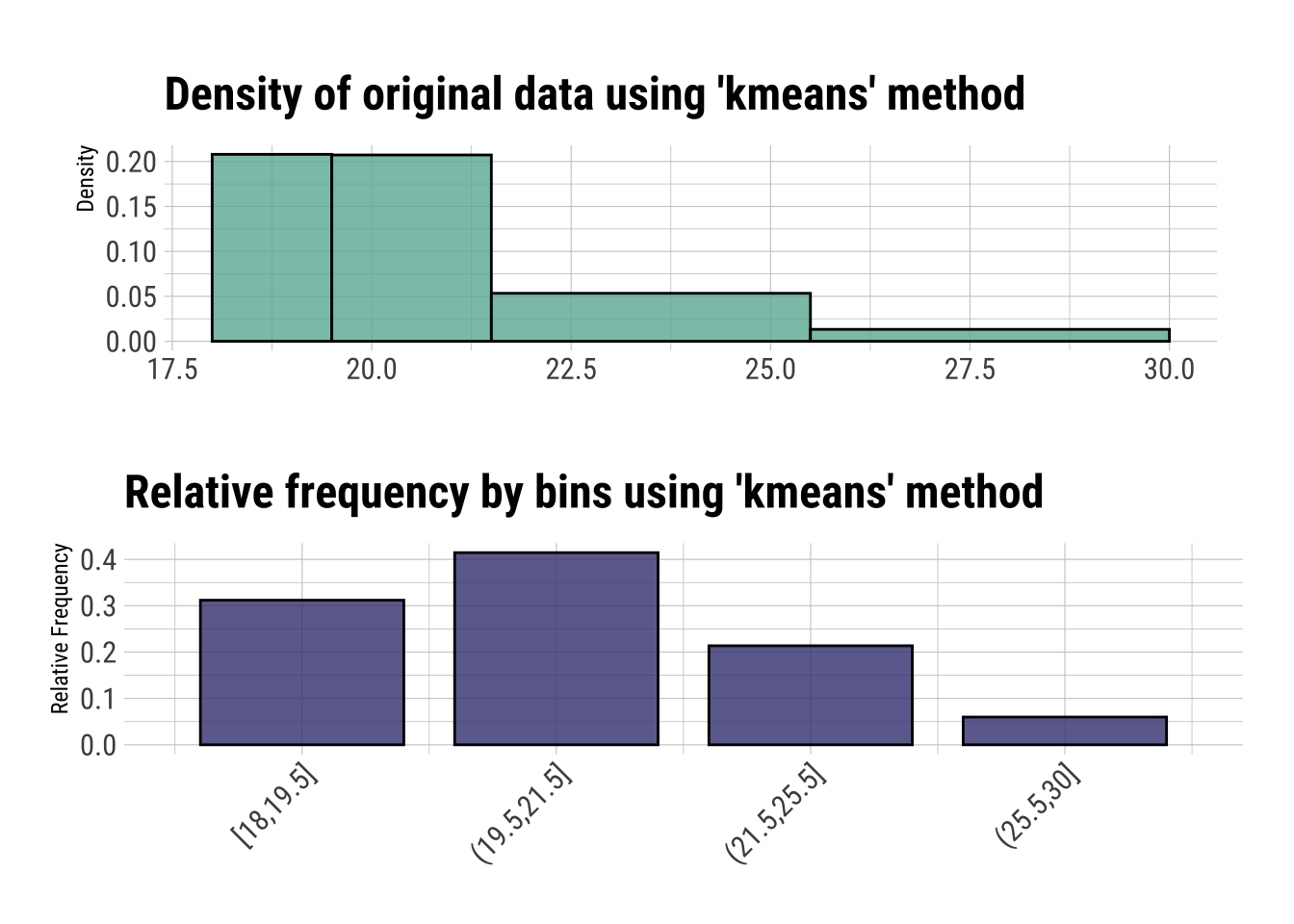
plot(binning(dfm_filtrado$Edad_imp, type = "bclust", nbins = 4))Error in bclust(x = var, centers = n, ...) :
Could not find valid cluster solution in 20 replications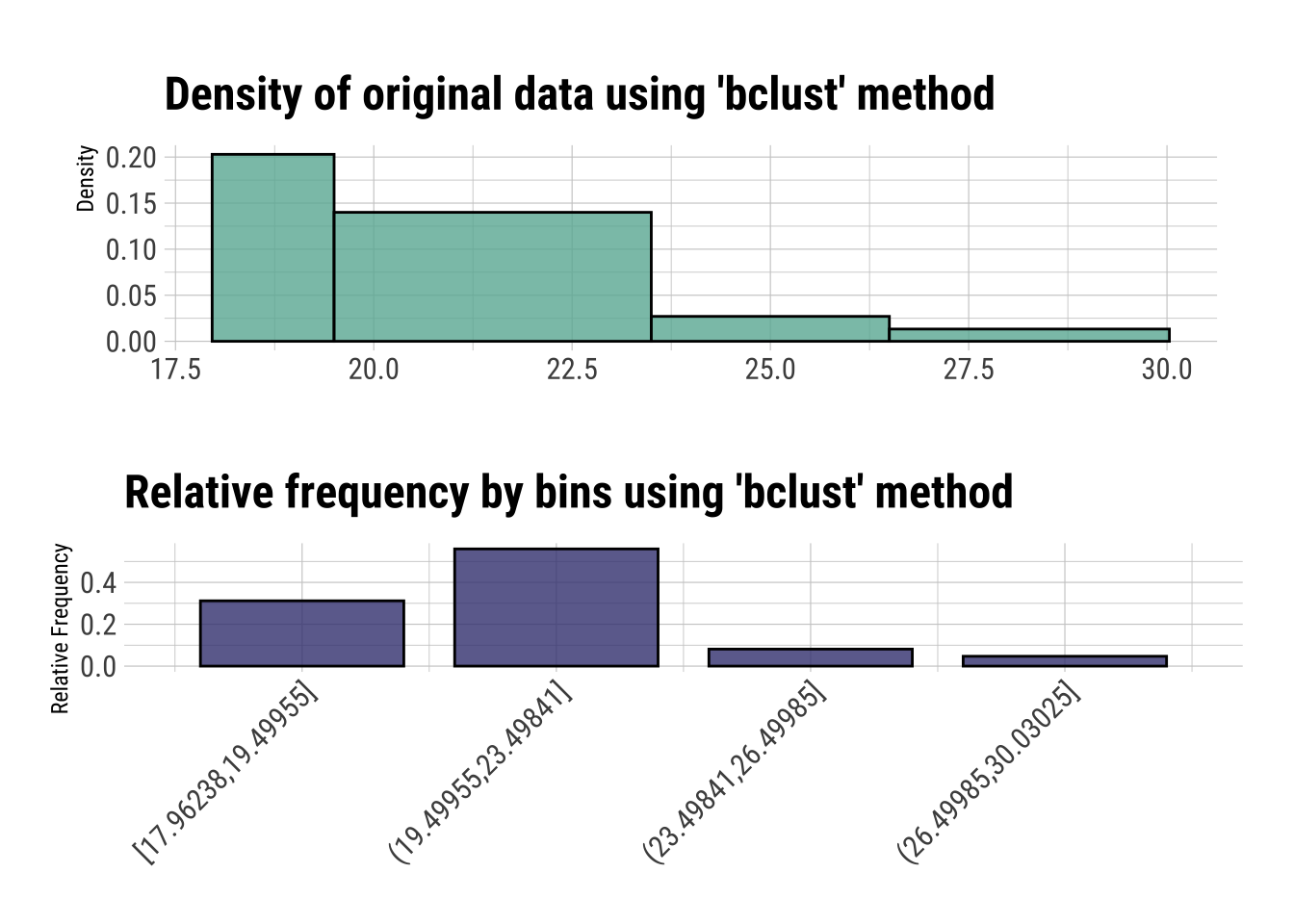
# Realizmos la agrupación
Edad_imp_cat <- (binning(dfm_filtrado$Edad_imp, nbins = 4, labels = c("18.5", "19.5", "21", ">22")))
summary(Edad_imp_cat) levels freq rate
1 18.5 73 0.3119658
2 19.5 56 0.2393162
3 21 60 0.2564103
4 >22 45 0.1923077dfm_filtrado$Edad_imp_cat <- Edad_imp_cat # Añadimos al dataset4.4.1 Finalización de fase de reparación
Finalizamos creado un dataframe “reparado-preparado” al que llamamos df.
# Eliminar la clase "imputation" de todas las columnas que la tengan
df <- dfm_filtrado %>%
mutate(across(everything(), ~ if ("imputation" %in% class(.)) unclass(.) else .))
df <- select(df, -Edad, -Estudios, -Curso, -V1, -V2, -V3, -V8, -V25, -Edad_imp, -Estudios_num)5 Exploración
5.1 Análisis exploratorio
describe(df) %>%
flextable()described_variables | n | na | mean | sd | se_mean | IQR | skewness | kurtosis | p00 | p01 | p05 | p10 | p20 | p25 | p30 | p40 | p50 | p60 | p70 | p75 | p80 | p90 | p95 | p99 | p100 |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
N | 234 | 0 | 123.739316 | 70.025290 | 4.57769639 | 119.50 | -0.027602884 | -1.1804054361 | 1 | 3.33 | 12.65 | 26.6 | 52.6 | 64.25 | 75.9 | 101.2 | 124.5 | 147.80 | 172.1 | 183.75 | 195.8 | 219.70 | 231.35 | 240.67 | 243 |
V4 | 234 | 0 | 3.910256 | 1.155534 | 0.07553963 | 2.00 | -0.766118686 | 0.4780778260 | 1 | 1.00 | 1.00 | 2.0 | 3.0 | 3.00 | 4.0 | 4.0 | 4.0 | 4.00 | 5.0 | 5.00 | 5.0 | 5.00 | 5.00 | 6.00 | 6 |
V5 | 234 | 0 | 2.658120 | 1.442235 | 0.09428184 | 2.00 | 0.658620834 | -0.2652769923 | 1 | 1.00 | 1.00 | 1.0 | 1.0 | 1.00 | 2.0 | 2.0 | 3.0 | 3.00 | 3.0 | 3.00 | 4.0 | 5.00 | 6.00 | 6.00 | 6 |
V6 | 234 | 0 | 3.047009 | 1.274516 | 0.08331769 | 2.00 | 0.400619147 | -0.2519240190 | 1 | 1.00 | 1.00 | 1.3 | 2.0 | 2.00 | 2.0 | 3.0 | 3.0 | 3.00 | 4.0 | 4.00 | 4.0 | 5.00 | 5.35 | 6.00 | 6 |
V7 | 234 | 0 | 3.243590 | 1.244930 | 0.08138364 | 2.00 | 0.080152906 | -0.3425049319 | 1 | 1.00 | 1.00 | 2.0 | 2.0 | 2.00 | 3.0 | 3.0 | 3.0 | 4.00 | 4.0 | 4.00 | 4.0 | 5.00 | 5.00 | 6.00 | 6 |
V9 | 234 | 0 | 3.012821 | 1.334523 | 0.08724048 | 2.00 | 0.358878340 | -0.3614489787 | 1 | 1.00 | 1.00 | 1.0 | 2.0 | 2.00 | 2.0 | 3.0 | 3.0 | 3.00 | 4.0 | 4.00 | 4.0 | 5.00 | 6.00 | 6.00 | 6 |
V10 | 234 | 0 | 3.034188 | 1.263089 | 0.08257068 | 2.00 | 0.231713064 | -0.3284358486 | 1 | 1.00 | 1.00 | 1.0 | 2.0 | 2.00 | 2.0 | 3.0 | 3.0 | 3.00 | 4.0 | 4.00 | 4.0 | 5.00 | 5.00 | 6.00 | 6 |
V11 | 234 | 0 | 3.115385 | 1.228916 | 0.08033675 | 2.00 | 0.197868652 | -0.3982602206 | 1 | 1.00 | 1.00 | 2.0 | 2.0 | 2.00 | 2.0 | 3.0 | 3.0 | 3.00 | 4.0 | 4.00 | 4.0 | 5.00 | 5.00 | 6.00 | 6 |
V12 | 234 | 0 | 3.089744 | 1.258645 | 0.08228017 | 2.00 | 0.181179035 | -0.4293692879 | 1 | 1.00 | 1.00 | 1.0 | 2.0 | 2.00 | 2.0 | 3.0 | 3.0 | 3.00 | 4.0 | 4.00 | 4.0 | 5.00 | 5.00 | 6.00 | 6 |
V13 | 234 | 0 | 3.051282 | 1.289417 | 0.08429179 | 2.00 | 0.364215590 | -0.2336716058 | 1 | 1.00 | 1.00 | 1.0 | 2.0 | 2.00 | 2.0 | 3.0 | 3.0 | 3.00 | 4.0 | 4.00 | 4.0 | 5.00 | 5.35 | 6.00 | 6 |
V14 | 234 | 0 | 3.435897 | 1.238749 | 0.08097958 | 1.00 | -0.357380072 | -0.3541814311 | 1 | 1.00 | 1.00 | 2.0 | 2.0 | 3.00 | 3.0 | 3.0 | 4.0 | 4.00 | 4.0 | 4.00 | 4.0 | 5.00 | 5.00 | 6.00 | 6 |
V15 | 234 | 0 | 3.811966 | 1.263089 | 0.08257068 | 2.00 | -0.607402956 | -0.0004991714 | 1 | 1.00 | 1.00 | 2.0 | 3.0 | 3.00 | 3.0 | 4.0 | 4.0 | 4.00 | 5.0 | 5.00 | 5.0 | 5.00 | 6.00 | 6.00 | 6 |
V16 | 234 | 0 | 3.427350 | 1.238275 | 0.08094860 | 1.00 | -0.050830510 | -0.5162898627 | 1 | 1.00 | 1.00 | 2.0 | 2.0 | 3.00 | 3.0 | 3.0 | 3.5 | 4.00 | 4.0 | 4.00 | 4.0 | 5.00 | 5.00 | 6.00 | 6 |
V17 | 234 | 0 | 4.034188 | 1.276608 | 0.08345446 | 2.00 | -0.825926645 | 0.2035026124 | 1 | 1.00 | 1.00 | 2.0 | 3.0 | 3.00 | 4.0 | 4.0 | 4.0 | 5.00 | 5.0 | 5.00 | 5.0 | 5.00 | 6.00 | 6.00 | 6 |
V18 | 234 | 0 | 3.222222 | 1.215855 | 0.07948290 | 1.00 | 0.346332621 | -0.1260630508 | 1 | 1.00 | 1.00 | 2.0 | 2.0 | 3.00 | 3.0 | 3.0 | 3.0 | 3.00 | 4.0 | 4.00 | 4.0 | 5.00 | 5.00 | 6.00 | 6 |
V19 | 234 | 0 | 2.790598 | 1.085882 | 0.07098630 | 1.00 | 0.404826010 | 0.2694972529 | 1 | 1.00 | 1.00 | 1.0 | 2.0 | 2.00 | 2.0 | 3.0 | 3.0 | 3.00 | 3.0 | 3.00 | 4.0 | 4.00 | 5.00 | 6.00 | 6 |
V20 | 234 | 0 | 2.705128 | 1.128358 | 0.07376309 | 1.00 | 0.492432674 | 0.1374002090 | 1 | 1.00 | 1.00 | 1.0 | 2.0 | 2.00 | 2.0 | 2.0 | 3.0 | 3.00 | 3.0 | 3.00 | 4.0 | 4.00 | 5.00 | 6.00 | 6 |
V21 | 234 | 0 | 3.153846 | 1.176416 | 0.07690473 | 2.00 | 0.591107622 | 0.1644267528 | 1 | 1.00 | 1.65 | 2.0 | 2.0 | 2.00 | 3.0 | 3.0 | 3.0 | 3.00 | 3.1 | 4.00 | 4.0 | 5.00 | 5.35 | 6.00 | 6 |
V22 | 234 | 0 | 2.799145 | 1.103175 | 0.07211680 | 1.00 | 0.328104307 | 0.1536101413 | 1 | 1.00 | 1.00 | 1.0 | 2.0 | 2.00 | 2.0 | 3.0 | 3.0 | 3.00 | 3.0 | 3.00 | 4.0 | 4.00 | 5.00 | 6.00 | 6 |
V23 | 234 | 0 | 3.243590 | 1.278940 | 0.08360694 | 2.00 | 0.229919620 | -0.3820108917 | 1 | 1.00 | 1.00 | 2.0 | 2.0 | 2.00 | 3.0 | 3.0 | 3.0 | 3.00 | 4.0 | 4.00 | 4.0 | 5.00 | 5.35 | 6.00 | 6 |
V24 | 234 | 0 | 3.166667 | 1.229990 | 0.08040697 | 2.00 | 0.236507946 | -0.1908285242 | 1 | 1.00 | 1.00 | 2.0 | 2.0 | 2.00 | 3.0 | 3.0 | 3.0 | 3.00 | 4.0 | 4.00 | 4.0 | 5.00 | 5.00 | 6.00 | 6 |
V26 | 234 | 0 | 3.299145 | 1.220462 | 0.07978409 | 2.00 | 0.281631270 | -0.5097864314 | 1 | 1.00 | 2.00 | 2.0 | 2.0 | 2.00 | 3.0 | 3.0 | 3.0 | 3.80 | 4.0 | 4.00 | 4.0 | 5.00 | 5.00 | 6.00 | 6 |
V27 | 234 | 0 | 2.042735 | 1.200094 | 0.07845261 | 2.00 | 1.134450285 | 0.7886710997 | 1 | 1.00 | 1.00 | 1.0 | 1.0 | 1.00 | 1.0 | 1.0 | 2.0 | 2.00 | 3.0 | 3.00 | 3.0 | 4.00 | 4.35 | 5.67 | 6 |
V28 | 234 | 0 | 3.529915 | 1.161416 | 0.07592413 | 1.00 | 0.009776589 | -0.2979449826 | 1 | 1.00 | 2.00 | 2.0 | 3.0 | 3.00 | 3.0 | 3.0 | 3.0 | 4.00 | 4.0 | 4.00 | 5.0 | 5.00 | 5.00 | 6.00 | 6 |
V29 | 234 | 0 | 3.824786 | 1.239230 | 0.08101103 | 2.00 | -0.208491374 | -0.3600970164 | 1 | 1.00 | 2.00 | 2.0 | 3.0 | 3.00 | 3.0 | 4.0 | 4.0 | 4.00 | 5.0 | 5.00 | 5.0 | 5.00 | 6.00 | 6.00 | 6 |
V30 | 234 | 0 | 2.418803 | 1.353674 | 0.08849247 | 2.00 | 0.859265896 | -0.0453129281 | 1 | 1.00 | 1.00 | 1.0 | 1.0 | 1.00 | 1.0 | 2.0 | 2.0 | 2.00 | 3.0 | 3.00 | 3.0 | 5.00 | 5.00 | 6.00 | 6 |
V31 | 234 | 0 | 3.162393 | 1.286825 | 0.08412239 | 2.00 | 0.400263068 | -0.2464726043 | 1 | 1.00 | 1.00 | 2.0 | 2.0 | 2.00 | 2.9 | 3.0 | 3.0 | 3.00 | 4.0 | 4.00 | 4.0 | 5.00 | 6.00 | 6.00 | 6 |
V32 | 234 | 0 | 2.888889 | 1.529553 | 0.09999001 | 2.00 | 0.624235933 | -0.4185539868 | 1 | 1.00 | 1.00 | 1.0 | 1.0 | 2.00 | 2.0 | 2.0 | 3.0 | 3.00 | 3.0 | 4.00 | 4.0 | 6.00 | 6.00 | 6.00 | 6 |
V33 | 234 | 0 | 2.884615 | 1.329566 | 0.08691643 | 2.00 | 0.335087726 | -0.5727894440 | 1 | 1.00 | 1.00 | 1.0 | 2.0 | 2.00 | 2.0 | 3.0 | 3.0 | 3.00 | 3.0 | 4.00 | 4.0 | 5.00 | 5.00 | 6.00 | 6 |
Estudios_imp | 234 | 0 | 3.256410 | 1.553917 | 0.10158274 | 3.75 | -0.296181554 | -1.3212063019 | 1 | 1.00 | 1.00 | 1.0 | 1.0 | 1.25 | 3.0 | 3.0 | 3.0 | 4.00 | 5.0 | 5.00 | 5.0 | 5.00 | 5.00 | 5.00 | 5 |
V1_imp | 234 | 0 | 3.529915 | 1.142790 | 0.07470650 | 1.00 | -0.396105175 | -0.0739802243 | 1 | 1.00 | 1.00 | 2.0 | 3.0 | 3.00 | 3.0 | 3.0 | 4.0 | 4.00 | 4.0 | 4.00 | 4.0 | 5.00 | 5.00 | 6.00 | 6 |
V2_imp | 234 | 0 | 3.298291 | 1.068189 | 0.06982968 | 1.15 | -0.182698164 | -0.2351917728 | 1 | 1.00 | 1.26 | 2.0 | 2.0 | 2.85 | 3.0 | 3.0 | 3.2 | 4.00 | 4.0 | 4.00 | 4.0 | 4.88 | 5.00 | 5.67 | 6 |
V3_imp | 234 | 0 | 3.271795 | 1.108209 | 0.07244592 | 1.20 | -0.045163396 | -0.2859936429 | 1 | 1.00 | 1.00 | 2.0 | 2.0 | 2.80 | 3.0 | 3.0 | 3.0 | 3.96 | 4.0 | 4.00 | 4.0 | 5.00 | 5.00 | 6.00 | 6 |
V25_imp | 234 | 0 | 2.636752 | 1.225838 | 0.08013551 | 1.00 | 0.314342112 | -0.7570215607 | 1 | 1.00 | 1.00 | 1.0 | 1.0 | 2.00 | 2.0 | 2.0 | 3.0 | 3.00 | 3.0 | 3.00 | 4.0 | 4.00 | 5.00 | 5.00 | 5 |
df %>%
group_by(Estudios_imp) %>%
describe(V2_imp) %>%
flextable()described_variables | Estudios_imp | n | na | mean | sd | se_mean | IQR | skewness | kurtosis | p00 | p01 | p05 | p10 | p20 | p25 | p30 | p40 | p50 | p60 | p70 | p75 | p80 | p90 | p95 | p99 | p100 |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
V2_imp | 1 | 59 | 0 | 3.111864 | 1.2013046 | 0.15639653 | 2.0 | 0.01856741 | -0.4308981 | 1 | 1.000 | 1.00 | 1.48 | 2 | 2.0 | 2.80 | 3.00 | 3.0 | 3.16 | 4.0 | 4.0 | 4.0 | 5.00 | 5.0 | 5.42 | 6 |
V2_imp | 2 | 3 | 0 | 3.333333 | 0.5773503 | 0.33333333 | 0.5 | 1.73205081 | 3 | 3.000 | 3.00 | 3.00 | 3 | 3.0 | 3.00 | 3.00 | 3.0 | 3.20 | 3.4 | 3.5 | 3.6 | 3.80 | 3.9 | 3.98 | 4 | |
V2_imp | 3 | 70 | 0 | 3.151429 | 1.0951465 | 0.13089504 | 2.0 | 0.10934420 | -0.2525548 | 1 | 1.000 | 1.36 | 2.00 | 2 | 2.0 | 2.74 | 3.00 | 3.0 | 3.28 | 4.0 | 4.0 | 4.0 | 4.64 | 5.0 | 5.31 | 6 |
V2_imp | 4 | 23 | 0 | 3.434783 | 1.2294155 | 0.25635085 | 1.5 | -0.39175917 | -0.8424457 | 1 | 1.088 | 1.46 | 2.00 | 2 | 2.5 | 3.00 | 3.16 | 3.8 | 4.00 | 4.0 | 4.0 | 4.6 | 5.00 | 5.0 | 5.00 | 5 |
V2_imp | 5 | 79 | 0 | 3.526582 | 0.8576517 | 0.09649335 | 1.0 | -0.38722656 | 0.7397921 | 1 | 1.780 | 2.00 | 2.00 | 3 | 3.0 | 3.00 | 3.44 | 4.0 | 4.00 | 4.0 | 4.0 | 4.0 | 4.20 | 5.0 | 5.22 | 6 |
df %>%
normality() %>% # Comprueba normalidad con la prueba Shapiro-Wilk
flextable()vars | statistic | p_value | sample |
|---|---|---|---|
N | 0.9562546 | 0.0000015188396544191486 | 234 |
V4 | 0.8838249 | 0.0000000000020561624408 | 234 |
V5 | 0.8863687 | 0.0000000000029524807158 | 234 |
V6 | 0.9240341 | 0.0000000013562470984343 | 234 |
V7 | 0.9335932 | 0.0000000086901068838914 | 234 |
V9 | 0.9251814 | 0.0000000016814542823103 | 234 |
V10 | 0.9288132 | 0.0000000033668690742636 | 234 |
V11 | 0.9317405 | 0.0000000059884263505804 | 234 |
V12 | 0.9325104 | 0.0000000069853214680845 | 234 |
V13 | 0.9251769 | 0.0000000016800176075574 | 234 |
V14 | 0.9148418 | 0.0000000002598978874113 | 234 |
V15 | 0.9027486 | 0.0000000000349325953484 | 234 |
V16 | 0.9355735 | 0.0000000130301779862719 | 234 |
V17 | 0.8796562 | 0.0000000000011495727107 | 234 |
V18 | 0.9168904 | 0.0000000003717624647757 | 234 |
V19 | 0.9100079 | 0.0000000001141077887080 | 234 |
V20 | 0.9102824 | 0.0000000001194754629023 | 234 |
V21 | 0.9012280 | 0.0000000000274642760359 | 234 |
V22 | 0.9120669 | 0.0000000001614450483737 | 234 |
V23 | 0.9299915 | 0.0000000042375170032360 | 234 |
V24 | 0.9281714 | 0.0000000029734293345160 | 234 |
V26 | 0.9276923 | 0.0000000027111953519232 | 234 |
V27 | 0.8086474 | 0.0000000000000002971002 | 234 |
V28 | 0.9306750 | 0.0000000048476597145570 | 234 |
V29 | 0.9320748 | 0.0000000064016871603610 | 234 |
V30 | 0.8615008 | 0.0000000000001061045524 | 234 |
V31 | 0.9219612 | 0.0000000009244951178979 | 234 |
V32 | 0.8892367 | 0.0000000000044690584518 | 234 |
V33 | 0.9150422 | 0.0000000002690973234912 | 234 |
Estudios_imp | 0.8218141 | 0.0000000000000011391060 | 234 |
V1_imp | 0.9140676 | 0.0000000002273442330857 | 234 |
V2_imp | 0.9453435 | 0.0000001082077535757260 | 234 |
V3_imp | 0.9436924 | 0.0000000745473529992983 | 234 |
V25_imp | 0.9001024 | 0.0000000000230207256856 | 234 |
df %>%
group_by(Universidad) %>%
normality() %>%
flextable()variable | Universidad | statistic | p_value | sample |
|---|---|---|---|---|
N | A | 0.96403561 | 0.0013801983535960782138730 | 133 |
N | B | 0.74757203 | 0.0000000010147688865577820 | 71 |
N | C | 0.95208548 | 0.1922070823775794412568985 | 30 |
V4 | A | 0.87683783 | 0.0000000040517988477208329 | 133 |
V4 | B | 0.90808749 | 0.0000733303888859193885594 | 71 |
V4 | C | 0.84453684 | 0.0004774794082551656764814 | 30 |
V5 | A | 0.89275354 | 0.0000000245453697247782444 | 133 |
V5 | B | 0.86102155 | 0.0000013229729061312137156 | 71 |
V5 | C | 0.90784437 | 0.0131348506524232785941386 | 30 |
V6 | A | 0.91991747 | 0.0000008068591282111012834 | 133 |
V6 | B | 0.91141374 | 0.0001010269995723580492260 | 71 |
V6 | C | 0.92714958 | 0.0412692913871410965720443 | 30 |
V7 | A | 0.93029878 | 0.0000036655459335979467769 | 133 |
V7 | B | 0.93024142 | 0.0006957244101149058978714 | 71 |
V7 | C | 0.91759760 | 0.0232460443012000132767536 | 30 |
V9 | A | 0.92847674 | 0.0000027865004839023955259 | 133 |
V9 | B | 0.90684191 | 0.0000651337693270724398757 | 71 |
V9 | C | 0.91099386 | 0.0157657526358473236427482 | 30 |
V10 | A | 0.93061135 | 0.0000038435723939834569714 | 133 |
V10 | B | 0.91754130 | 0.0001851215576853444970320 | 71 |
V10 | C | 0.93537086 | 0.0683031092485538171565906 | 30 |
V11 | A | 0.93271519 | 0.0000053048781005453905283 | 133 |
V11 | B | 0.91891519 | 0.0002126493718918261752009 | 71 |
V11 | C | 0.89379116 | 0.0059403351249021551713780 | 30 |
V12 | A | 0.93342004 | 0.0000059167074841396478588 | 133 |
V12 | B | 0.91661757 | 0.0001687468553632372441752 | 71 |
V12 | C | 0.92329131 | 0.0326754168170818592997762 | 30 |
V13 | A | 0.91374636 | 0.0000003455599987164156738 | 133 |
V13 | B | 0.92132451 | 0.0002718804218567774893915 | 71 |
V13 | C | 0.89886348 | 0.0078787770807759883312515 | 30 |
V14 | A | 0.92844202 | 0.0000027720806380172883290 | 133 |
V14 | B | 0.90595926 | 0.0000599145004169910575529 | 71 |
V14 | C | 0.80037974 | 0.0000666133571913921670830 | 30 |
V15 | A | 0.91714101 | 0.0000005484950127019236364 | 133 |
V15 | B | 0.88283836 | 0.0000076300221300262455518 | 71 |
V15 | C | 0.85800903 | 0.0009164795678727015448839 | 30 |
V16 | A | 0.93730188 | 0.0000109146774825635125216 | 133 |
V16 | B | 0.91112647 | 0.0000982474766428872786032 | 71 |
V16 | C | 0.90273370 | 0.0098030029193141420690294 | 30 |
V17 | A | 0.90151392 | 0.0000000710133575519791580 | 133 |
V17 | B | 0.86866841 | 0.0000023995991182504615515 | 71 |
V17 | C | 0.80423052 | 0.0000783703779972319140900 | 30 |
V18 | A | 0.91080211 | 0.0000002334285331896464338 | 133 |
V18 | B | 0.89170395 | 0.0000163606225813222936726 | 71 |
V18 | C | 0.89615424 | 0.0067718337013128364690329 | 30 |
V19 | A | 0.90370978 | 0.0000000935028123675656526 | 133 |
V19 | B | 0.90541585 | 0.0000569223098408791513387 | 71 |
V19 | C | 0.89248784 | 0.0055285666158467715314284 | 30 |
V20 | A | 0.91885508 | 0.0000006954629505603885433 | 133 |
V20 | B | 0.87743018 | 0.0000048644474041019310703 | 71 |
V20 | C | 0.87666993 | 0.0023662219192597147392199 | 30 |
V21 | A | 0.89786670 | 0.0000000453272731786488675 | 133 |
V21 | B | 0.90850558 | 0.0000763201458783843922828 | 71 |
V21 | C | 0.84186648 | 0.0004208781738922896917439 | 30 |
V22 | A | 0.90856488 | 0.0000001741303086676216716 | 133 |
V22 | B | 0.89059111 | 0.0000148404533498080437153 | 71 |
V22 | C | 0.90703679 | 0.0125376019585292739627613 | 30 |
V23 | A | 0.93028211 | 0.0000036562990903583990405 | 133 |
V23 | B | 0.91922942 | 0.0002195331476472392158786 | 71 |
V23 | C | 0.89776124 | 0.0074069625342661040215408 | 30 |
V24 | A | 0.92469579 | 0.0000015963296711694815773 | 133 |
V24 | B | 0.92956647 | 0.0006468464284930047162453 | 71 |
V24 | C | 0.91584932 | 0.0209602396966064759287196 | 30 |
V26 | A | 0.92618961 | 0.0000019856146138435563618 | 133 |
V26 | B | 0.91243494 | 0.0001115992657697208466404 | 71 |
V26 | C | 0.90448198 | 0.0108293607153130393844220 | 30 |
V27 | A | 0.83103015 | 0.0000000000462747128350288 | 133 |
V27 | B | 0.76603500 | 0.0000000027693812518796358 | 71 |
V27 | C | 0.77771930 | 0.0000264145048459567141847 | 30 |
V28 | A | 0.92528377 | 0.0000017390040887077237682 | 133 |
V28 | B | 0.91190246 | 0.0001059481981528283294674 | 71 |
V28 | C | 0.88340607 | 0.0033787362714365670388061 | 30 |
V29 | A | 0.92756960 | 0.0000024343665977837403132 | 133 |
V29 | B | 0.92233676 | 0.0003017519108420465396257 | 71 |
V29 | C | 0.84231931 | 0.0004299507924210789651387 | 30 |
V30 | A | 0.85421262 | 0.0000000003967882478891847 | 133 |
V30 | B | 0.86846633 | 0.0000023615503061902756999 | 71 |
V30 | C | 0.84027187 | 0.0003905139015234841671186 | 30 |
V31 | A | 0.92373879 | 0.0000013898012126415984136 | 133 |
V31 | B | 0.90420224 | 0.0000507948910778440075532 | 71 |
V31 | C | 0.93778946 | 0.0793164847164503500343002 | 30 |
V32 | A | 0.89451521 | 0.0000000302594792836326719 | 133 |
V32 | B | 0.86862525 | 0.0000023914183618488444260 | 71 |
V32 | C | 0.89304144 | 0.0056996580581570224083299 | 30 |
V33 | A | 0.91964187 | 0.0000007762704009931059733 | 133 |
V33 | B | 0.89873400 | 0.0000306724519060052927522 | 71 |
V33 | C | 0.88677153 | 0.0040484369794329538860822 | 30 |
Estudios_imp | A | 0.71674065 | 0.0000000000000106517473927 | 133 |
Estudios_imp | B | 0.09713472 | 0.0000000000000000009004821 | 71 |
Estudios_imp | C | 30 | ||
V1_imp | A | 0.91516607 | 0.0000004186753201931156286 | 133 |
V1_imp | B | 0.92502515 | 0.0003991801504789449614626 | 71 |
V1_imp | C | 0.74499075 | 0.0000075657957325261555068 | 30 |
V2_imp | A | 0.94373228 | 0.0000314659854631234553023 | 133 |
V2_imp | B | 0.95222485 | 0.0088320482971255276360978 | 71 |
V2_imp | C | 0.86405058 | 0.0012385000466245349795558 | 30 |
V3_imp | A | 0.94395921 | 0.0000326991678017866731990 | 133 |
V3_imp | B | 0.93313883 | 0.0009543173170494910354314 | 71 |
V3_imp | C | 0.92400514 | 0.0341124137131022242552802 | 30 |
V25_imp | A | 0.90590276 | 0.0000001235323962377279273 | 133 |
V25_imp | B | 0.87073274 | 0.0000028274426872482041304 | 71 |
V25_imp | C | 0.90905881 | 0.0140899960735791345695089 | 30 |
df %>%
normality(V15) %>%
flextable()vars | statistic | p_value | sample |
|---|---|---|---|
V15 | 0.9027486 | 0.0000000000349326 | 234 |
df %>%
plot_normality(V15)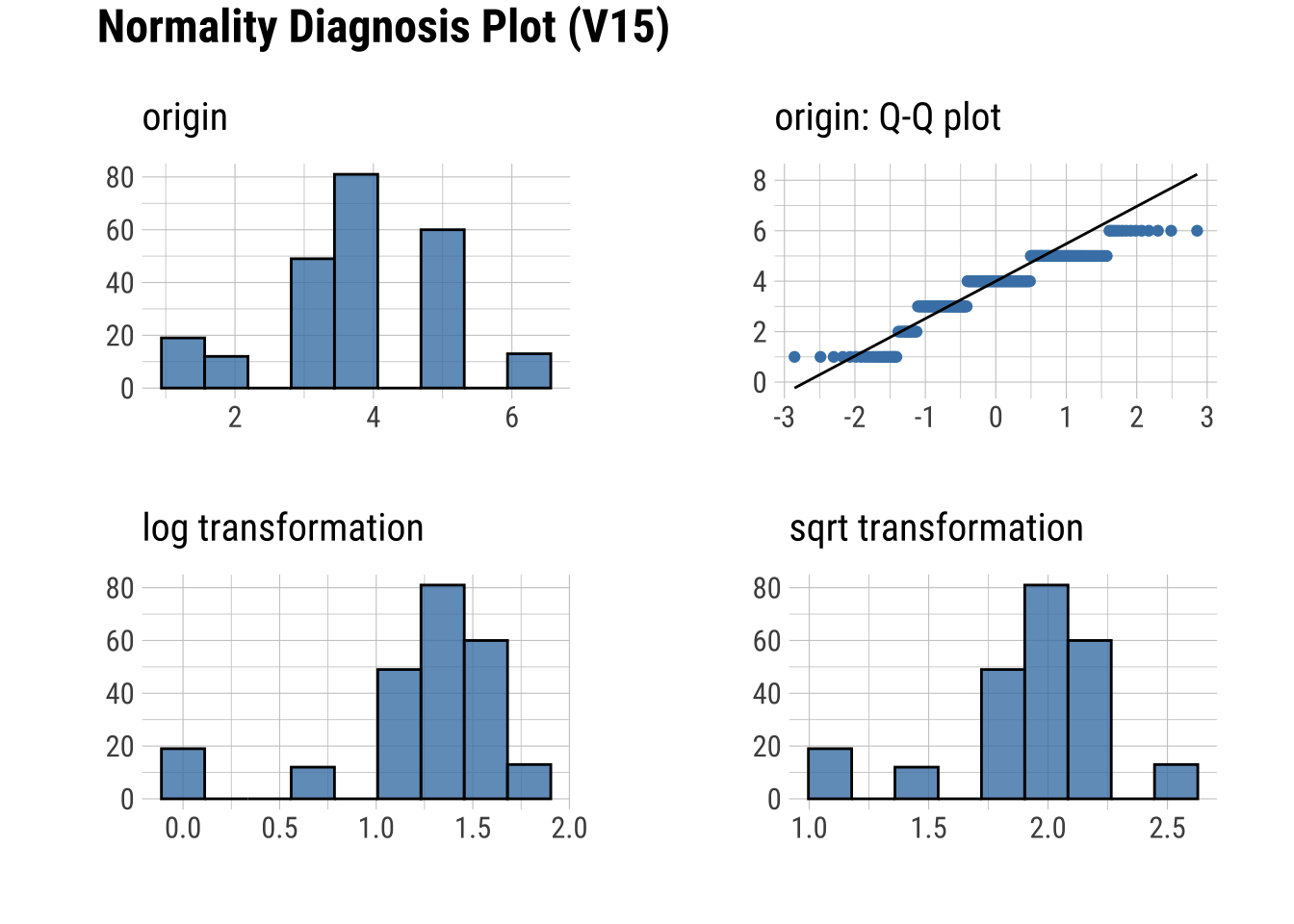
# Correlaciones
dfc <- select(df, V15, V20, V25_imp, V33) # Seleccionamos sólo algunas a modo de ejemplo
summary(correlate(dfc, method = "spearman"))* correlation type : generic
* variable type : numeric
* correlation method : spearman
* Matrix of Correlation
V15 V20 V25_imp V33
V15 1.000000000 0.04169035 0.004279304 -0.07248045
V20 0.041690354 1.00000000 0.606086067 -0.01747726
V25_imp 0.004279304 0.60608607 1.000000000 0.03089746
V33 -0.072480451 -0.01747726 0.030897460 1.00000000plot_correlate(dfc)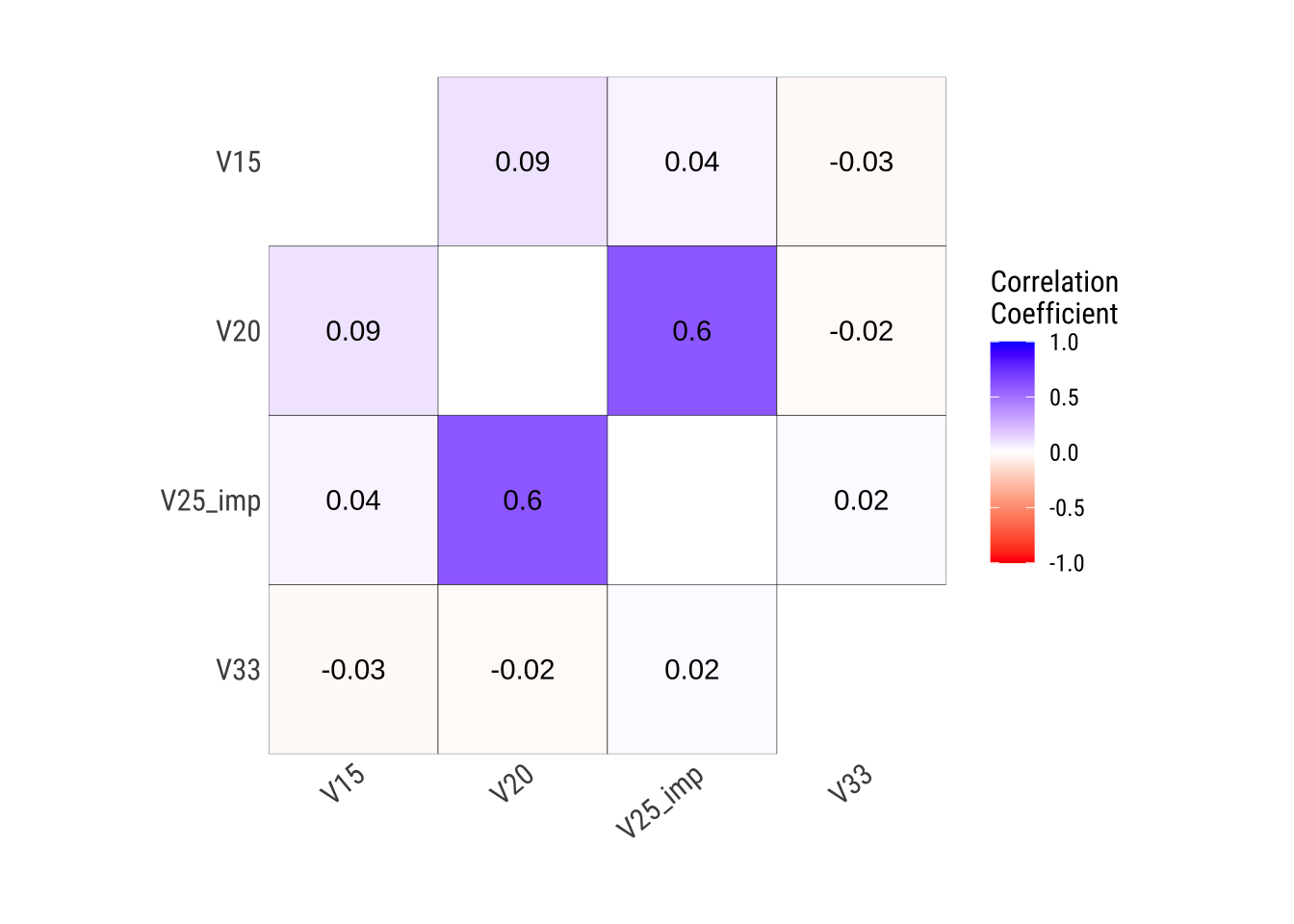
6 Informes
La función
diagnose_reportgenera un informe que combina la mayoría de las funciones de diagnóstico.La función
eda_web_reportgenera un informe con análisis exploratorio de datos.
Más información sobre los informes en:
# Informe diagnóstico. Combina la mayoría de las funciones de diagnóstico
diagnose_report(df)
diagnose_web_report(df)
# Informe EDA. Genera un informe con análisis exploratorio de datos.
eda_report(df)
eda_web_report(df)